ในช่วงที่การค้นคว้ายาใหม่ต้องเผชิญกับต้นทุนสูงและระยะเวลาการพัฒนาเป็นปีหรือทศวรรษ สตาร์ทอัพไบโอไทยกำลังนำเทคโนโลยีปัญญาประดิษฐ์ชั้นสูงมาเป็นตัวเร่งความเร็ว ด้วยการผสานวิธีการ Active Learning เข้ากับ Foundation Models เพื่อคัดกรองโมเลกุลนำร่องแบบไฮบริดระหว่างงานคอมพิวติ้งกับการทดลองจริง ผลลัพธ์จากโครงการนำร่องเบื้องต้นชี้ว่าแนวทางนี้ช่วยลดจำนวนการทดลองเชิงกายภาพและต้นทุนการทดลองได้อย่างมีนัยสำคัญ — ในบางกรณีลดการทดสอบทางห้องปฏิบัติการลงได้เกือบครึ่งหนึ่ง ขณะเดียวกันยังเพิ่มอัตราการพบ “hit” ของโมเลกุลที่มีศักยภาพ ซึ่งทำให้วงจรการค้นพบสารนำร่อง (lead discovery) สั้นลงและมีประสิทธิภาพมากขึ้น
บทความนี้จะพาผู้อ่านสำรวจกรณีศึกษาจากสตาร์ทอัพไทย กระบวนการทำงานตั้งแต่การเตรียมข้อมูล การออกแบบวงทดลองแบบ Active Learning การใช้งาน Foundation Models ในการคัดกรองโมเลกุล ไปจนถึงดัชนีชี้วัดผลลัพธ์ (เช่น อัตราการพบสารนำร่อง, การลดจำนวนการทดลองจริง, ต้นทุนต่อผู้สมัครสาร) พร้อมการวิเคราะห์ความท้าทายด้านคุณภาพข้อมูล ความโปร่งใสของโมเดล กฎระเบียบ และข้อเสนอเชิงนโยบายที่จำเป็นเพื่อขับเคลื่อนระบบนิเวศไบโอเทคของประเทศให้พร้อมรองรับการประยุกต์ใช้งาน AI ในวงการเภสัชกรรมและไบโอเทคอย่างปลอดภัยและยั่งยืน
บทนำ: ปัญหาโครงสร้างของการค้นคว้ายาและโอกาสจาก AI
บทนำ: ปัญหาทางโครงสร้างของการค้นคว้ายาและโอกาสจาก AI
การค้นคว้าและพัฒนายาแบบดั้งเดิมเป็นกระบวนการที่ใช้เวลาและทรัพยากรมหาศาล โดยภาพรวมการพัฒนายาจนถึงการอนุมัติและออกสู่ตลาดมักกินเวลาประมาณ 10–15 ปี และต้นทุนรวมที่งานวิจัยหลายชิ้นประเมินอยู่ในช่วงตั้งแต่หลายร้อยล้านดอลลาร์ไปจนถึงกว่าพันล้านดอลลาร์สหรัฐฯ ต่อยา หนึ่งในสาเหตุสำคัญที่ทำให้ต้นทุนและเวลาสูงคืออัตราการล้มเหลวที่สูงในแต่ละขั้นตอนการพัฒนา ทั้งจากความไม่แน่นอนทางชีวภาพ ปัญหาความเป็นพิษ และประสิทธิภาพที่ไม่เป็นไปตามคาดในขั้นคลินิก
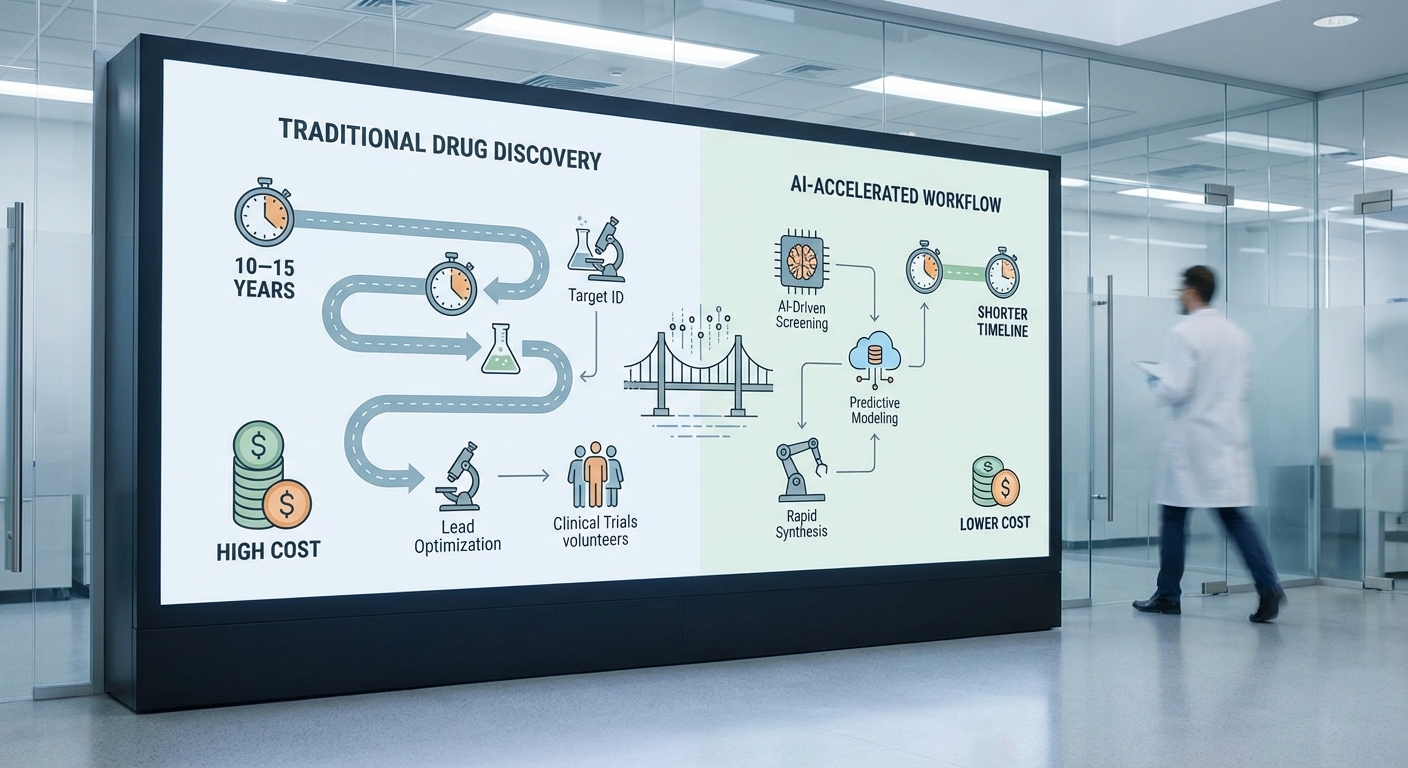
ในทางปฏิบัติ ห้องปฏิบัติการวิจัยเผชิญกับคอขวดหลายประการที่ส่งผลโดยตรงต่อ time-to-test และต้นทุน ได้แก่:
- เวลาในการเตรียมและรันการทดลอง — การสังเคราะห์โมเลกุล การเตรียมตัวอย่าง และการรัน assay แต่ละครั้งอาจใช้เวลาหลายวันถึงหลายสัปดาห์
- ต้นทุนสารเคมีและ reagent — สารเคมีเฉพาะทางและ reagent บางชนิดมีราคาสูง โดยเฉพาะเมื่อทดลองในสเกลที่ต้องการความแม่นยำสูง
- throughput ที่จำกัด — แม้จะมีระบบอัตโนมัติ แต่การทดสอบเชิงชีวภาพบางชนิดยังต้องพึ่งการปฏิบัติงานแบบแมนนวลหรือเครื่องมือเฉพาะ ส่งผลให้จำนวนตัวอย่างที่ทดสอบได้ต่อเวลาไม่เพียงพอต่อความต้องการ
- อัตราการล้มเหลวซ้ำซ้อน — ผลการทดลองในห้องปฏิบัติการอาจไม่สอดคล้องกับผลในโมเดลต่อไป ทำให้ต้องทำซ้ำหลายรอบและเพิ่มต้นทุนการทดลองจริง
ในบริบทนี้ เทคโนโลยีปัญญาประดิษฐ์โดยเฉพาะ Active Learning และ Foundation Models เปิดช่องทางใหม่ในการลดจำนวนและความถี่ของการทดลองทางห้องปฏิบัติการ โดยแนวคิดสำคัญคือการย้ายภาระการตัดสินใจอันดับต้นมาไว้ที่การคำนวณ คัดกรอง และจัดลำดับความสำคัญของโมเลกุลที่มีแนวโน้มสูงที่สุดก่อนการทดลองจริง:
Active Learning เป็นกรอบการเรียนรู้เชิงโต้ตอบที่โมเดลจะเลือกตัวอย่างที่มีประโยชน์สูงสุดสำหรับการทดลองถัดไป ทำให้สามารถลดจำนวนการทดลองจริงลงอย่างมีประสิทธิภาพ ตัวอย่างจากงานวิจัยหลายกรณีศึกษาแสดงว่าแนวทางนี้สามารถลดปริมาณการทดลอง wet-lab ได้อย่างมีนัยสำคัญ (มีการรายงานการลดได้หลายเท่าในบริบทต่าง ๆ) ขณะที่ Foundation Models — โมเดลขนาดใหญ่ที่ผ่านการฝึกบนข้อมูลขนาดใหญ่เช่นโครงสร้างโมเลกุล ลำดับโปรตีน และข้อมูลการทดลองเชิงชีวภาพ — ช่วยสร้างตัวแทนเชิงคุณลักษณะ (embeddings) ที่มีพลังสำหรับการทำนายคุณสมบัติทางเคมี ช่วยให้การคัดกรองเชิงคอมพิวเตอร์ (in silico screening) มีความแม่นยำและครอบคลุมมากขึ้น
สำหรับสตาร์ทอัพไบโอในไทย ซึ่งมักเผชิญข้อจำกัดด้านงบประมาณและเวลา การผสานกันของ Active Learning และ Foundation Models เป็นโอกาสเชิงกลยุทธ์ที่สำคัญ เพราะช่วยให้ทีมสามารถ:
- มุ่งทรัพยากรทดลองไปยังโมเลกุลที่มีความน่าจะเป็นสำเร็จสูงสุด
- ลดค่าใช้จ่ายด้าน reagent และการสังเคราะห์โดยหลีกเลี่ยงการทดสอบตัวอย่างที่มีข้อมูลบ่งชี้ความเป็นไปได้ต่ำ
- เร่งเวลาไปสู่การทดสอบทางคลินิกขั้นต้น (IND-enabling studies) และการนำผลงานเชิงพาณิชย์มาใช้จริง
แม้การประยุกต์ใช้ AI จะไม่สามารถแทนที่การทดลองทางชีววิทยาและการยืนยันเชิงประจักษ์ได้ทั้งหมด แต่การนำ Active Learning และ Foundation Models มาเป็นส่วนหนึ่งของเวิร์กโฟลว์การค้นคว้ายาช่วยเปิดทางให้สตาร์ทอัพไบโอไทยทำงานได้เร็วขึ้น ถูกกว่าด้วยงบประมาณที่จำกัด และมีโอกาสแข่งขันในเวทีสากลมากขึ้น โดยยังต้องผสมผสานการวางแผนเชิงการทดลองและการตรวจสอบคุณภาพข้อมูลอย่างรัดกุมเพื่อให้ได้ผลลัพธ์ที่น่าเชื่อถือและสามารถนำไปสู่การพัฒนาเชิงคลินิกได้จริง
Active Learning และ Foundation Models: พื้นฐานเชิงเทคนิคที่ควรรู้
Active Learning และ Foundation Models: พื้นฐานเชิงเทคนิคที่ควรรู้
ในบริบทการค้นคว้ายาและการคัดกรองโมเลกุลเชิงชีวเคมี สตาร์ทอัพที่ผสานแนวทาง Active Learning กับ Foundation Models มักจะสร้างวงจรป้อนกลับที่ช่วยลดจำนวนการทดลองจริง (wet‑lab) ที่จำเป็นลงอย่างมีนัยสำคัญ โดยใช้โมเดลพรีเทรนบนข้อมูลขนาดใหญ่เพื่อเป็นฐานความรู้ แล้วใช้กลไก Active Learning ในการเลือกตัวอย่างที่มีค่าสำหรับการทดลองเพิ่มเติม การผสมผสานนี้ทำให้สามารถคัดกรองคลังสารขนาดใหญ่ (เช่น 10^5–10^7 สาร) ให้เหลือตัวอย่างนำร่องเพียงหลักร้อยถึงหลักพันสำหรับการทดสอบจริงได้อย่างมีประสิทธิภาพ
Active Learning ทำงานเป็นวงจรป้อนกลับแบบเลือกตัวอย่างที่ “คุ้มค่า” ต่อการทดลองจริง โดยทั่วไปประกอบด้วยขั้นตอนหลักคือ (1) เริ่มจากชุดข้อมูลเล็กๆ ที่มีป้ายกำกับ (labeled set) และชุดข้อมูลไม่ป้ายกำกับขนาดใหญ่ (unlabeled pool) (2) ฝึกโมเดล (surrogate model) บน labeled set (3) ประเมินความไม่แน่นอนหรือเกณฑ์การได้ประโยชน์ (acquisition function) บน pool เพื่อเลือกตัวอย่างที่จะส่งไปทดลองจริง (4) เอาผลการทดลองกลับมาเป็นป้ายกำกับใหม่แล้วอัปเดตโมเดลซ้ำ กระบวนการนี้ช่วยให้ทรัพยากรการทดลองถูกใช้กับตัวอย่างที่ให้ข้อมูลมากที่สุด แทนที่จะสุ่มหรือไล่ตรวจทุกตัวอย่าง
เทคนิคการเลือกตัวอย่างที่นิยมมีหลายรูปแบบ เช่น
- Uncertainty sampling — เลือกตัวอย่างที่โมเดลคาดการณ์ด้วยความไม่แน่นอนสูง (เช่น entropy, margin confidence) เพื่อให้การทดลองช่วยลดความไม่แน่นอนได้มากที่สุด
- Query‑by‑committee — ใช้กลุ่มโมเดล (ensemble) หลายตัวแล้วเลือกตัวอย่างที่คำตอบระหว่างโมเดลขัดแย้งกันมากที่สุด ซึ่งบ่งชี้ว่าตัวอย่างนั้นให้ข้อมูลเรียนรู้สูง
- Expected model change / Expected improvement — เลือกตัวอย่างที่คาดว่าจะเปลี่ยนพารามิเตอร์ของโมเดลได้มากที่สุด หรือคาดว่าจะปรับปรุงสมรรถนะของงานเป้าหมายได้มากที่สุด
- Bayesian approaches — ใช้ Gaussian processes หรือวิธี Bayesian เพื่อวัดความไม่แน่นอนเชิงปริมาณและคำนวณ acquisition function เช่น BALD
ในฝั่งของ Foundation Models แนวคิดหลักคือการพรีเทรนโมเดลขนาดใหญ่บนข้อมูลที่หลากหลายและปริมาณมาก จากนั้นนำ representations ที่ได้ไปปรับต่อ (fine‑tune) กับงานเฉพาะทางในชีวเคมีหรือเคมี ตัวอย่างในงานเคมีเชิงคำนวณได้แก่
- SMILES‑based transformers — โมเดลภาษาที่เรียนรู้จากลำดับ SMILES (เช่น SMILES transformers) โดยใช้ objective เช่น masked language modeling เพื่อได้ embedding ที่จับลักษณะทางเคมี
- ChemBERTa, MolBERT — โมเดล BERT‑style ที่พรีเทรนบนคลังสารขนาดใหญ่ (เช่น PubChem, ZINC) เพื่อเป็นตัวแทนของโมเลกุลสำหรับงานพยากรณ์สมบัติ (property prediction) และการคัดกรอง
- Graph Neural Networks (GNNs) — โมเดลที่ทำงานบนโครงสร้างกราฟของโมเลกุล (atom nodes, bond edges) เช่น message‑passing networks, Graphormer ซึ่งถูกใช้ทั้งเป็น foundation model และเป็น surrogate model ใน active learning
- Structure predictors — สำหรับโปรตีนและเป้าหมายทางชีวภาพ โมเดลอย่าง AlphaFold ให้การทำนายโครงสร้าง 3 มิติที่มีความละเอียดสูง ทำให้การจำลองการจับกันของโมเลกุล (docking) และการออกแบบเป้าหมายมีพื้นฐานจากโครงสร้างที่ดีขึ้น
- Equivariant / 3D neural networks เช่น SchNet, DimeNet, SE(3)‑Transformers — ใช้ข้อมูลเชิงทรงกลม (3D) ในการทำนายพลังงานและสมบัติที่เกี่ยวข้องกับโครงสร้าง
ตัวอย่างการประยุกต์ใช้งานร่วมกันในกระบวนการ virtual screening และการออกแบบโมเลกุลมีดังนี้:
- เริ่มด้วย foundation model เพื่อสร้าง embedding ของห้องสมุดโมเลกุลขนาดใหญ่ จากนั้นฝึก surrogate model แบบ light‑weight (เช่น GNN หรือมิใช่เชิงลึกเท่านั้น) บนชุดข้อมูลทดลองเล็กๆ
- ใช้ Active Learning (uncertainty sampling หรือ query‑by‑committee) เพื่อเลือกกลุ่มโมเลกุลที่จะส่งไปทดสอบ in vitro/in vivo รอบแรก — เทคนิคนี้ช่วยลดจำนวนการทดสอบจริงจากล้านรายการไปสู่หลักพันหรือหลักร้อยสำหรับการทดสอบนำร่อง ขึ้นกับความซับซ้อนของปัญหา
- ผลการทดลองถูกป้อนกลับมาเพื่ออัปเดต surrogate model และปรับกลยุทธ์การคัดเลือกในรอบถัดไป ซึ่งมักเพิ่มอัตราการได้สารนำร่องที่มีสมบัติที่ต้องการ (enrichment) เมื่อเทียบกับการสุ่ม
- ในงานออกแบบโมเลกุล (de novo design) generative foundation models สามารถผลิตข้อเสนอสารใหม่แล้วใช้ surrogate model + acquisition function เพื่อคัดเลือกสารที่ควรสังเคราะห์จริง
- หากมีเป้าหมายโปรตีน โมเดลโครงสร้างเช่น AlphaFold ทำให้สามารถนำโครงสร้างเป้าหมายมาปรับปรุงการคำนวณ docking หรือการคาดการณ์ binding affinity ได้ดีขึ้น เพิ่มความแม่นยำให้กับ surrogate model
ข้อควรระวังเชิงเทคนิคที่ผู้บริหารควรทราบคือการตั้งค่า acquisition function ที่ไม่ดีอาจนำไปสู่การเลือกตัวอย่างที่ไม่หลากหลาย (redundancy) หรือเกิดปัญหา domain shift เมื่อ pool ของสารมีการกระจายต่างจากข้อมูลพรีเทรน อีกทั้ง foundation models ต้องการทรัพยากรพรีเทรนและการดูแลเรื่องการปรับเทียบ (calibration) ของความไม่แน่นอน หากจัดการดี โมเดลชุดนี้สามารถลดต้นทุนการทดลองจริงได้อย่างมีนัยสำคัญ—งานวิจัยหลายชิ้นรายงานการลดจำนวนการทดลองที่จำเป็นได้หลายเท่าตัว (ขึ้นกับงานและการตั้งค่า อาจอยู่ในช่วงประมาณ 2–10×)
สรุปคือ การรวม Active Learning กับ Foundation Models ให้ทั้งประสิทธิภาพในการค้นหา (search efficiency) และความสามารถในการถ่ายโอนความรู้ (transferability) ซึ่งเป็นรากฐานเชิงเทคนิคที่สำคัญสำหรับสตาร์ทอัพไบโอที่ต้องการเร่งกระบวนการค้นยาและลดต้นทุนการทดลองจริงในเชิงปฏิบัติ
กรณีศึกษานำร่องของสตาร์ทอัพไบโอไทย: ขั้นตอนและผลลัพธ์เชิงตัวอย่าง
กรณีศึกษานำร่องของสตาร์ทอัพไบโอไทย: ขั้นตอนและผลลัพธ์เชิงตัวอย่าง
ในกรณีศึกษานำร่องนี้ ทีมงานสตาร์ทอัพไบโอไทยได้ออกแบบกระบวนการผสมผสาน Foundation Models กับกลไก Active Learning เพื่อคัดกรองโมเลกุลนำร่องสำหรับโปรแกรมค้นหายา เป้าหมายเชิงธุรกิจคือการลดจำนวนการทดลองในห้องปฏิบัติการ (wet‑lab) ที่มีต้นทุนสูง พร้อมเพิ่มอัตราการพบสารออกฤทธิ์ ("hit rate") ให้สูงกว่าวิธีดั้งเดิม กระบวนการนำร่องถูกแบ่งเป็นหลายขั้นตอนชัดเจน ตั้งแต่การรวบรวมข้อมูลจนถึงการเลือกตัวอย่างสำหรับทดลองจริง โดยในตัวอย่างสมมติที่ใกล้เคียงสถานการณ์จริง ทีมงานเริ่มจากชุดโมเลกุลขนาดหมื่นรายการและลดลงเหลือเพียงร้อยกว่ารายการสำหรับการทดลอง wet‑lab
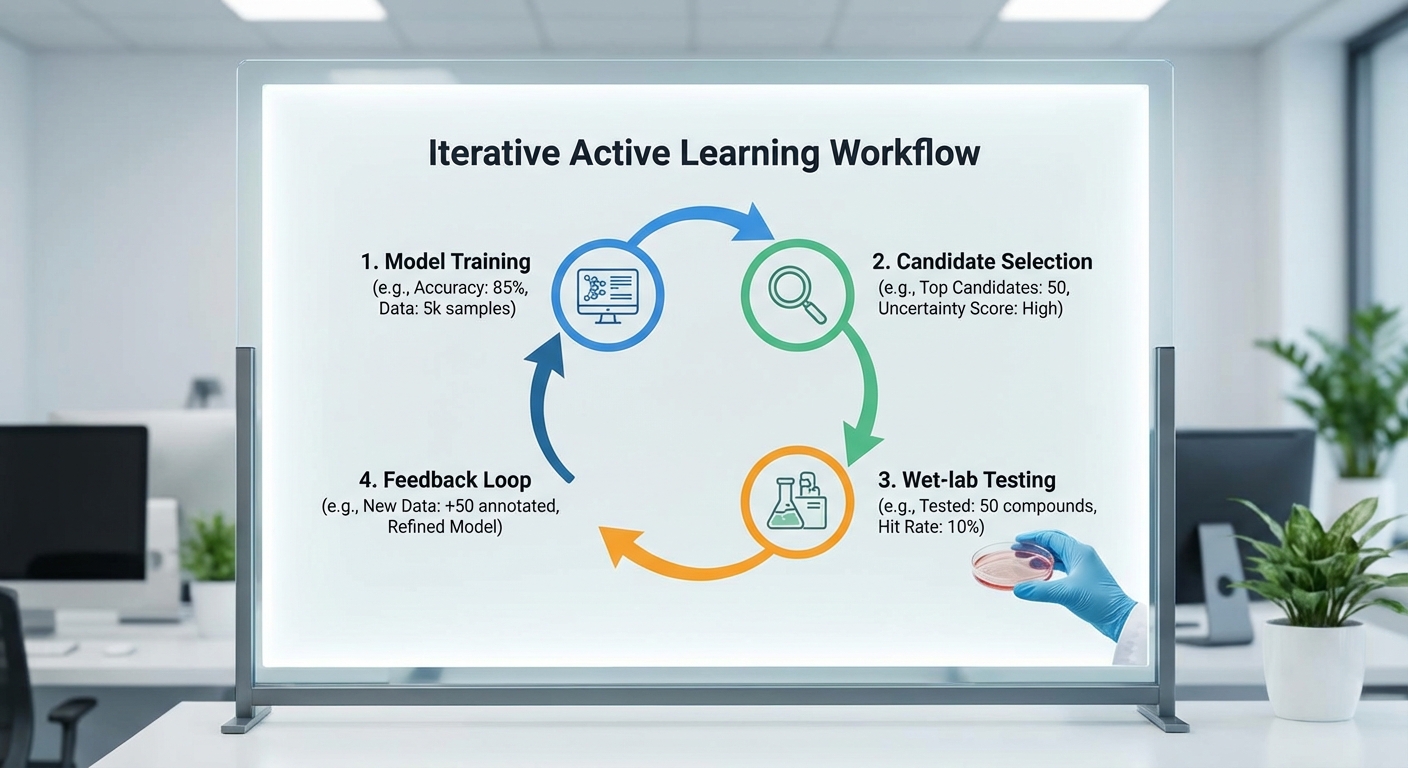
ขั้นตอนนำร่องที่ใช้ในเคสตัวอย่างประกอบด้วย:
- รวบรวมข้อมูลโมเลกุล: นำเข้าโมเลกุลจากฐานข้อมูลสาธารณะ (เช่น PubChem/ChEMBL) และชุดข้อมูลภายในบริษัท รวมทั้งข้อมูลไบโอแอสเซย์ที่มีฉลากผลการทดสอบ
- พรีโปรเซส: ทำความสะอาดโครงสร้างโมเลกุล (standardization), สกัดฟีเจอร์เชิงเคมี (fingerprints, graph representations), และจัดการความไม่สมดุลของคลาสด้วยการสุ่มตัวอย่างหรือการถ่วงน้ำหนัก
- พรีเทรน/ฟายน์ทูน Foundation Model: ใช้โมเดลภาษาเชิงเคมีหรือกราฟ (pretrained on large chemical corpora) แล้วฟายน์ทูนด้วยข้อมูลแอสเซย์ของโปรเจกต์เพื่อให้โมเดลเรียนรู้สัญญาณเฉพาะของเป้าหมายโปรตีน
- Active Learning loop: เริ่มจากการคัดกรองเชิงคอมพิวเตอร์เพื่อจัดอันดับโมเลกุล จากนั้นสุ่ม/เลือกโมเลกุลที่มีความไม่แน่นอนสูงและคาดว่าจะให้ข้อมูลมากที่สุดส่งเข้า wet‑lab ผลการทดลองจะถูกป้อนกลับมาปรับปรุงโมเดลในรอบถัดไป จนกว่าจะบรรลุเกณฑ์
- เลือกตัวอย่างสำหรับ wet‑lab: หลังหลายรอบ Active Learning เลือกกลุ่มสุดท้ายขนาดเล็ก (ร้อยกว่ารายการ) สำหรับการทดสอบเชิงประสิทธิผลและพิษวิทยาเบื้องต้น
ตัวอย่างเชิงตัวเลขจาก pilot สมมติ: ทีมเริ่มด้วยชุดโมเลกุลประมาณ 25,000 รายการ ผ่านการพรีโปรเซสและให้คะแนนโดย Foundation Model ก่อนการทำ Active Learning รอบแรก ระบบคัดกรองเชิงคอมพิวเตอร์ลดจำนวนลงเหลือ ~1,200 รายการ ที่มีคะแนนสูงสุดและ/หรือมีความไม่แน่นอนเชิงนโยบาย จากนั้นทำ Active Learning ประมาณ 6–8 รอบ โดยในแต่ละรอบเลือกส่ง 50–200 ตัวอย่างไปทดสอบ wet‑lab ซึ่งท้ายที่สุด pipeline ได้กลุ่มตัวอย่างเชิงทดลองจริงประมาณ 120–160 รายการ สำหรับการทดสอบเชิงลึก
ผลลัพธ์เชิงตัวอย่างที่น่าสังเกต ได้แก่:
- การเพิ่มอัตรา hit rate: ในตัวอย่างสมมติ เคยพบอัตรา hit rate ประมาณ 0.5% ด้วยการคัดกรองแบบดั้งเดิม (random/heuristic screening) แต่หลังใช้ Foundation Models + Active Learning พบอัตรา hit rate ประมาณ 5–8% (เพิ่มขึ้น 10–16 เท่า)
- การลดจำนวนการทดลอง wet‑lab: จากเดิมถ้าต้องการค้นหา hit หลายตัวอาจต้องทำการทดลองหลายพันรายการ แต่ในเคสนี้ลดการทดลองจริงลงกว่า 90% (จากหลักพันเหลือหลักร้อย) ช่วยลดทั้งเวลาและต้นทุน
- ผลประหยัดต้นทุนและเวลา: คำนวณเชิงตัวอย่างพบว่าต้นทุนการเตรียมและรันการทดลองลดลงประมาณ 60–75% เมื่อเทียบกับเวิร์กโฟลว์ดั้งเดิม และระยะเวลาในการค้นหาเบื้องต้น (lead identification) ลดจากประมาณ 12–18 เดือน เหลือเพียง 3–6 เดือน ในโปรเจกต์นี้
นอกจากตัวเลขเชิงปริมาณ ทีมงานยังสังเกตข้อดีเชิงคุณภาพ เช่น การค้นพบเคมีสเปซที่หลากหลายขึ้น (diversity) เนื่องจากกลไก Active Learning สนับสนุนการเลือกตัวอย่างที่ไม่ซ้ำและให้ข้อมูลมากที่สุด รวมทั้งลดความเสี่ยงจากการยึดติดกับ heuristic เดิม ๆ ซึ่งช่วยเพิ่มโอกาสค้นพบ scaffold ใหม่ ๆ ผลลัพธ์เหล่านี้ยังชี้ให้เห็นว่าการลงทุนในงานข้อมูล (data curation, labeling) และการฟายน์ทูน foundation models ให้เหมาะสมกับปัญหาสามารถให้ผลตอบแทนเชิงเศรษฐศาสตร์ที่ชัดเจนสำหรับสตาร์ทอัพไบโอระดับกลาง
ข้อสรุปเชิงธุรกิจจากเคสตัวอย่างคือการผสาน Foundation Models กับ Active Learning เป็นแนวทางที่มีประสิทธิผลสำหรับสตาร์ทอัพที่มีทรัพยากรจำกัด ช่วยให้ทีมสามารถโฟกัสทรัพยากร wet‑lab ไปยังตัวอย่างที่มีโอกาสสำเร็จสูงสุด สร้างความได้เปรียบด้านต้นทุน เวลา และอัตราการสำเร็จของโครงการค้นหายาในระยะเริ่มต้น
กระบวนการเชิงปฏิบัติและมาตรวัดผล (metrics) ที่ใช้ประเมิน
ภาพรวมและกรอบการประเมิน
เมื่อสตาร์ทอัพไบโอไทยผสาน Active Learning กับ Foundation Models ในการคัดกรองโมเลกุลนำร่อง การวัดผลต้องครอบคลุมทั้งมิติทางเทคนิคและมิติทางเศรษฐศาสตร์ เพื่อยืนยันว่า loop ของ AI เพิ่มอัตราการค้นพบจริงและลดต้นทุนการทดลองเชิงปฏิบัติ (wet-lab). มาตรวัดที่เลือกควรสามารถตอบคำถามสำคัญได้ว่า “โมเดลช่วยค้นเจอโมเลกุลที่มีผลจริงได้ดีกว่าเดิมเท่าใด” และ “ลดจำนวนและค่าใช้จ่ายการทดลองจริงได้เท่าใด”
มาตรวัดหลักเชิงประสิทธิภาพ (Performance Metrics)
- Hit rate — สัดส่วนของโมเลกุลที่ทดสอบแล้วได้ผลตามเกณฑ์ (confirmed hits) ต่อจำนวนการทดสอบทั้งหมดในชุดตัวอย่างที่เลือก เช่น hit rate = confirmed hits / tested compounds. ควรคำนวณทั้งในแต่ละรอบของ Active Learning และสะสมตั้งแต่ต้นโครงการเพื่อจับแนวโน้ม
- Enrichment factor (EF) — วัดการเพิ่มขึ้นของความเข้มข้นของ hits เมื่อเทียบกับการสุ่มหรือ baseline: EF@x% = (hits_in_selected_subset / size_selected_subset) / (total_hits_in_library / library_size). ค่า EF มากกว่า 1 แสดงว่าเกิดการ “enrichment” ซึ่งสะท้อนประสิทธิภาพการคัดกรองของโมเดล
- Precision@k — ความแม่นยำของโมเดลในกลุ่มลำดับสูงสุด k ตัวอย่าง (top-k). ใช้เมื่อทีมเลือกทดลองเฉพาะ top-ranked candidates เช่น precision@100 = confirmed hits ใน top-100 / 100
- AUC-ROC — วัดความสามารถของโมเดลในการแยกแยะระหว่าง active vs inactive โดยรวม ใช้เป็นมาตรวัดเชิงภาพรวมของความสามารถในการจัดอันดับ แม้จะไม่สะท้อนต้นทุนโดยตรง แต่ช่วยในการเปรียบเทียบเวอร์ชันโมเดล
มาตรวัดเชิงเศรษฐศาสตร์และเวลา
- Cost-per-confirmed-hit (ค่าใช้จ่ายต่อการยืนยันโมเลกุลที่มีผล) — คำนวณจากผลรวมของค่าใช้จ่ายทางคอมพิวติ้ง (โมเดล, annotation, Active Learning orchestration) และค่าใช้จ่าย wet-lab ที่ใช้ในการยืนยัน หารด้วยจำนวน confirmed hits ในช่วงเวลาหรือรอบที่สนใจ: Cost-per-hit = (compute_costs + wetlab_costs) / confirmed_hits. มาตรวัดนี้เป็นตัวชี้วัดเชิงธุรกิจที่ตรงที่สุดสำหรับ ROI
- Time-to-next-test / Time-to-hit — เวลาที่ใช้ตั้งแต่มี hypothesis/compound ถูกเลือกจนถึงผลการทดสอบยืนยัน (รวมคิวในห้องปฏิบัติการและเวลาการเตรียมตัว). การลดเวลานี้ช่วยลดเวลาสู่การตัดสินใจและเพิ่มความเร็วของพอร์ตโฟลิโอ
- Reduction in wet-lab experiments — วัดเป็นจำนวนสัมบูรณ์และร้อยละของการลดการทดลองจริงเมื่อเปรียบเทียบ workflow แบบเดิมกับแบบผสาน AI: reduction% = (N_traditional − N_AI) / N_traditional × 100
ตัวอย่างการคำนวณเปรียบเทียบ (ตัวอย่างเชิงปฏิบัติ)
สมมติฐานเบื้องต้นสำหรับการเปรียบเทียบ:
- ขนาดไลบรารี (library_size) = 100,000 โมเลกุล
- Traditional workflow: ทดลองจริง 10,000 ตัวอย่าง (selected by historical criteria), ได้ confirmed hits = 20 → hit rate = 20/10,000 = 0.2%
- AI + Active Learning workflow: ทดลองจริง 2,000 ตัวอย่าง (prioritized by AL loop), ได้ confirmed hits = 40 → hit rate = 40/2,000 = 2.0%
คำนวณมาตรวัดสำคัญ:
- Enrichment factor (เทียบกับ traditional selection) = (2.0%) / (0.2%) = 10×
- Precision@200 (ตัวอย่าง top-200) — หาก top-200 อยู่ในชุดทดสอบ 2,000 และยืนยันได้ 20 hits ใน top-200 → precision@200 = 20/200 = 10%
- Reduction in wet-lab experiments = (10,000 − 2,000) / 10,000 × 100 = 80% ลดการทดลองจริงลงถึง 80%
- Cost-per-confirmed-hit (สมมติค่าใช้จ่าย) — สมมติ wet-lab cost ต่อการทดสอบ = 500 USD, compute & orchestration cost (รวมค่า annotation, model ops) = 20,000 USD สำหรับรอบ:
- Traditional: wet-lab_costs = 10,000 × 500 = 5,000,000 USD; total_cost = 5,000,000 + 0 = 5,000,000; cost-per-hit = 5,000,000 / 20 = 250,000 USD/confirmed hit
- AI+AL: wet-lab_costs = 2,000 × 500 = 1,000,000 USD; total_cost = 1,000,000 + 20,000 = 1,020,000 USD; cost-per-hit = 1,020,000 / 40 = 25,500 USD/confirmed hit
กระบวนการรายงานและ Retrospective Analysis เพื่อยืนยันประสิทธิภาพ
- เก็บข้อมูลเชิงทดลองและเมตาดาต้าอย่างเป็นระบบ — บันทึกผลการทดสอบทุกตัวอย่างพร้อมลำดับการคัดเลือก, เวอร์ชันโมเดล, เงื่อนไขการทดลอง และค่าใช้จ่ายที่เกี่ยวข้อง เพื่อให้สามารถย้อนกลับมาวิเคราะห์ได้
- การวิเคราะห์ย้อนหลัง (retrospective analysis) — ดำเนินการเปรียบเทียบแบบก่อน‑หลัง (pre-post) และ cross-validation ของผลลัพธ์: คำนวณ hit rate, EF, precision@k, AUC-ROC สำหรับแต่ละรอบ AL และสะสมเป็นช่วงเวลา พร้อมการสร้าง Confidence Intervals (เช่น bootstrap) เพื่อประเมินความแน่นอนทางสถิติ
- A/B testing และ statistical significance — เมื่อตั้งค่าเป็นระยะถี่ ควรรัน A/B test ระหว่าง pipeline แบบดั้งเดิมและ pipeline ที่ผสาน AI เพื่อยืนยันความเหนือกว่าเชิงสถิติ (ตัวอย่างเช่นทดสอบความแตกต่างของ hit rates ด้วย Fisher’s exact test หรือ chi-squared test ในระดับความเชื่อมั่น 95%)
- การวิเคราะห์ต้นทุนเชิงลึก — แยกต้นทุนเป็นหมวด (wet-lab per assay, setup costs, compute, human curation) และรายงานค่าใช้จ่ายต่อ hit พร้อม sensitivity analysis (เช่น หาก wet-lab cost ลด/เพิ่ม 20% ผลกระทบต่อ cost-per-hit เป็นอย่างไร)
- monitoring และ feedback loop — ติดตาม drift ของโมเดล (performance decay), monitor calibration (reliability diagram), และบันทึก learning curve ของ AL (performance ต่อจำนวนตัวอย่างที่ถูกทดลองจริง). รายงานควรมี dashboard สรุป KPI รายสัปดาห์/รายเดือนเพื่อการตัดสินใจเชิงธุรกิจ
สรุปแล้ว การวัดผลที่รัดกุมประกอบด้วยทั้งมาตรวัดเชิงประสิทธิภาพ เช่น hit rate, enrichment factor, precision@k และ AUC-ROC รวมกับมาตรวัดเชิงเศรษฐศาสตร์ เช่น cost-per-hit และ reduction in wet‑lab experiments. การรายงานต้องมีการทำ retrospective analysis และการทดสอบเชิงสถิติเพื่อยืนยันว่า Active Learning loop สร้างมูลค่าเชิงธุรกิจอย่างแท้จริงก่อนการขยายผลในระดับพอร์ตโฟลิโอ.
ความท้าทายด้านข้อมูล กฎระเบียบ และจริยธรรม
ความท้าทายด้านข้อมูล: คุณภาพข้อมูลและความครบถ้วนของ metadata
การนำ Active Learning ผสานกับ Foundation Models มาใช้คัดกรองโมเลกุลเพื่อเร่งการค้นยา ขึ้นอยู่กับคุณภาพของข้อมูลที่ป้อนเข้าอย่างยิ่ง — ทั้งความถูกต้อง ความสมบูรณ์ และความหลากหลายของตัวอย่างทดลอง ในภาคงาน Machine Learning อุตสาหกรรมมักประเมินว่า ประมาณ 60–80% ของเวลาและทรัพยากรถูกใช้ไปกับการจัดเตรียมและทำความสะอาดข้อมูล การละเลยรายละเอียดสำคัญ เช่น เงื่อนไขการทดลอง (assay protocol), batch identifier, วันที่เก็บตัวอย่าง, พารามิเตอร์การวัด และแหล่งที่มาของสารตัวอย่าง จะนำไปสู่ dataset shift หรือความเบี่ยงเบนเมื่อโมเดลถูกนำไปใช้กับข้อมูลที่แตกต่างจากข้อมูลฝึกสอน
ดังนั้นการมี metadata ที่ครบถ้วน ตามหลักการเช่น FAIR (Findable, Accessible, Interoperable, Reusable) รวมถึงการบันทึก provenance และ versioning ของ dataset เป็นสิ่งจำเป็น ไม่เพียงแต่ช่วยให้โมเดลมีประสิทธิภาพสูงขึ้น แต่ยังเป็นพื้นฐานสำหรับการตรวจสอบย้อนกลับ (audit trail) และการทำ validation แบบอิสระ เช่น การเปรียบเทียบผลข้ามห้องปฏิบัติการหรือ cross-site validation เพื่อจัดการกับปัญหา variability และ batch effects
- ปัญหาที่พบบ่อย: ป้ายกำกับผิดพลาด (label noise), ข้อมูลไม่สมดุล (class imbalance), ข้อมูลจาก assay ที่ไม่เป็นมาตรฐาน
- แนวทางบรรเทาความเสี่ยง: กำหนดมาตรฐาน metadata, ใช้ datasheets for datasets, ทำ data provenance และ pipeline ที่ reproducible
ความโปร่งใสของโมเดลและการอธิบายคำตัดสิน (explainability)
Foundation Models ขนาดใหญ่และโมเดลที่เรียนรู้ร่วมกับ Active Learning มักเป็นระบบที่ทำงานเชิงนามธรรมและยากต่อการอธิบายเหตุผลเบื้องหลังการคัดเลือกโมเลกุล การขาดความโปร่งใสจะสร้างปัญหาทางธุรกิจและกฎระเบียบเมื่อนำผลไปใช้ตัดสินใจเชิงคลินิกหรือเชิงการลงทุน ผู้กำกับดูแลและพันธมิตรทางการแพทย์มักต้องการหลักฐานเชิงเหตุผลที่ชัดเจนเมื่อยอมรับการใช้เทคโนโลยีใหม่
การจัดการกับประเด็นนี้ต้องผสานทั้งเทคนิคและการปกครอง เช่น การวัดความไม่แน่นอน (uncertainty quantification), การใช้โมดูล explainability (เช่น feature attribution, counterfactual explanations), การสร้าง model cards และรายงานการทดสอบความทนทาน (robustness tests) ที่ชัดเจน เพื่อให้ผู้เชี่ยวชาญสามารถตรวจสอบและท้าทายคำแนะนำของโมเดลได้
- ข้อเสนอปฏิบัติ: ระบุขอบเขตการใช้งานที่ปลอดภัยของโมเดล, บันทึกการตัดสินใจแบบมนุษย์ร่วม (human-in-the-loop), และทำ reporting ละเอียดสำหรับการประเมินภายนอก
- ความเสี่ยงถ้าไม่ได้ทำ: การตัดสินใจที่ไม่สามารถอธิบายได้อาจลดความเชื่อมั่นของนักลงทุนและหน่วยงานกำกับดูแล รวมถึงเพิ่มความเสี่ยงทางกฎหมาย
ความปลอดภัยทางชีวภาพ (biosafety) และความเสี่ยง dual-use
โมเดลที่สามารถเสนอหรือปรับแต่งโครงสร้างโมเลกุลมีศักยภาพทั้งในการช่วยค้นหาโมเลกุลที่เป็นประโยชน์และในทางกลับกัน อาจถูกนำไปใช้เพื่อสร้างสารพิษหรือเลือกออกแบบองค์ประกอบที่เป็นอันตราย (dual-use) นี่คือความเสี่ยงเชิงจริยธรรมและความปลอดภัยที่ต้องได้รับการจัดการอย่างเคร่งครัด
มาตรการที่จำเป็นครอบคลุมการทำ red-team testing, การตั้งค่า filters และ policy-based constraints ใน pipeline, การตรวจสอบโดยคณะกรรมการจริยธรรมทางชีวภาพ, และการประสานงานกับหน่วยงานที่เกี่ยวข้องเพื่อระบุพฤติกรรมที่อาจเป็นอันตราย นอกจากนี้ควรมีแผนการตอบสนองต่อเหตุการณ์ (incident response) และการทดสอบช่องโหว่ (penetration testing ในเชิงข้อมูล) เพื่อป้องกันการนำผลวิจัยไปใช้ในทางที่ไม่พึงประสงค์
ทรัพย์สินทางปัญญา (IP) และการปฏิบัติตามกฎระเบียบทั้งในไทยและระดับสากล
การใช้ข้อมูลจากแหล่งต่างๆ เพื่อฝึกสอน Foundation Models สร้างคำถามด้านสิทธิ์ของข้อมูล เช่น ใครเป็นเจ้าของข้อมูลที่ใช้ฝึกสอน, สิทธิ์ในการใช้โมเดลที่เกิดจากข้อมูลหลายแหล่ง, และความสามารถในการจดสิทธิบัตรสำหรับโมเลกุลที่ค้นพบโดยระบบอัตโนมัติ ปัญหาเหล่านี้มีผลต่อโมเดลธุรกิจ การเจรจาเชิงพาณิชย์ และการแบ่งผลประโยชน์กับสถาบันวิจัยหรือพันธมิตร
ในมิติของกฎระเบียบ บริษัทสตาร์ทอัพต้องปฏิบัติตามกฎข้อบังคับหลายรูปแบบ เช่น พระราชบัญญัติผลิตภัณฑ์สุขภาพของไทย (อย.), ข้อกำหนดด้านความปลอดภัยทางชีวภาพของหน่วยงานที่เกี่ยวข้อง, รวมถึงกฎคุ้มครองข้อมูลส่วนบุคคล (เช่น PDPA ในประเทศไทย และ GDPR หากเกี่ยวข้องกับข้อมูลสหภาพยุโรป) สำหรับการจำหน่ายหรือการทดลองทางคลินิกในต่างประเทศ ผู้ประกอบการต้องคำนึงถึงแนวทางของ FDA (สหรัฐฯ), EMA (ยุโรป) และแนวปฏิบัติระดับสากลเช่น Good Machine Learning Practice (GMLP) และคำแนะนำจาก WHO เรื่องเทคโนโลยีชีวภาพ
- ข้อปฏิบัติที่แนะนำ: ทำ MTA/DAA (Material Transfer Agreement / Data Access Agreement) ที่ชัดเจน, จัดทำ documentation ทางเทคนิคและการประเมินความเสี่ยง, และวางกลไกการแบ่งปันสิทธิผลประโยชน์อย่างเป็นธรรม
- การตรวจสอบภายในและภายนอก: จัดให้มี audit trail, third-party validation และการตรวจสอบตามข้อกำหนดของ อย. และหน่วยงานสากลก่อนการนำผลิตภัณฑ์ไปใช้จริง
สรุปคือ การนำ Active Learning และ Foundation Models มาใช้ในภาคไบโอเพื่อนำร่องการคัดกรองโมเลกุลนั้นให้ผลประโยชน์ด้านต้นทุนและความเร็วอย่างมาก แต่ต้องมีกรอบการจัดการข้อมูลที่เข้มแข็ง, นโยบายความโปร่งใสและการอธิบายคำตัดสินของโมเดล, มาตรการรักษาความปลอดภัยทางชีวภาพ และการปฏิบัติตามกฎระเบียบทั้งในประเทศและสากลเป็นพื้นฐาน เพื่อให้การนวัตกรรมนี้ยั่งยืนและรับผิดชอบต่อสังคม
ผลกระทบต่อระบบนิเวศสตาร์ทอัพไทยและเศรษฐกิจ
ผลกระทบต่อระบบนิเวศสตาร์ทอัพไทยและเศรษฐกิจ
การผสานวิธีการ Active Learning กับ Foundation Models ในการคัดกรองโมเลกุลนำร่องมีศักยภาพเปลี่ยนแปลงระบบนิเวศสตาร์ทอัพไบโอของไทยอย่างเป็นรูปธรรม โดยเฉพาะในด้านการเพิ่มความสามารถในการแข่งขันเชิงโลกและการลดอุปสรรคต้นทุน-เวลาให้กับการพัฒนายาและผลิตภัณฑ์ชีวภาพ นวัตกรรมเชิงคำนวณเหล่านี้ช่วยให้สตาร์ทอัพสามารถทดสอบสมมติฐานเชิงโมเลกุลก่อนลงทุนทดลองจริง ส่งผลให้ต้นทุนการทดลองในห้องปฏิบัติการและจำนวนการทดลองจริงลดลงอย่างมีนัยสำคัญ ซึ่งในหลายกรณีศึกษาทั่วโลกระบุว่าการใช้โมเดลเชิงคำนวณร่วมกับกลไกการเรียนรู้แบบแอกทีฟสามารถลดต้นทุนและจำนวนการทดลองจริงได้ราว 30–70% และย่นระยะเวลาขั้นตอนการค้นพบโมเลกุลนำร่อง (lead discovery) จากเดิมที่ใช้เวลาเป็นปีให้เหลือ ประมาณ 6–12 เดือน สำหรับโปรเจ็กต์นำร่องบางกรณี
ผลกระทบเชิงระบบจากการเปลี่ยนผ่านนี้ไม่ได้จำกัดเพียงการประหยัดต้นทุนเท่านั้น แต่ยังขยายไปสู่การส่งเสริมความร่วมมือข้ามศาสตร์ระหว่างวงการ AI, เคมี, และชีววิทยาอย่างเป็นรูปธรรม ความต้องการบุคลากรที่มีทักษะข้ามสาขาจะเพิ่มขึ้น ทั้งนักวิทยาศาสตร์ข้อมูลชีวภาพ (bioinformaticians), นักเคมีเชิงคำนวณ (computational chemists), และวิศวกร ML เฉพาะทาง ซึ่งคาดว่าจะสร้างงานเชิงเทคนิคและงานบริการรองรับหลายร้อยถึงหลักพันตำแหน่งภายใน 3–5 ปีแรกของการเติบโตของคลัสเตอร์ไบโอเทคในไทย นอกจากนี้ การทำงานร่วมกันระหว่างมหาวิทยาลัย โรงพยาบาล และภาคอุตสาหกรรมจะถูกตั้งเงื่อนไขใหม่ในแง่ของการแบ่งปันข้อมูล (data sharing), การออกแบบโครงการนำร่องร่วม (joint pilot), และกรอบการจัดการทรัพย์สินทางปัญญา (IP) ที่เอื้อต่อการต่อยอดเชิงพาณิชย์
ในมุมมองการเงินและการลงทุน การยกระดับความสามารถด้านคอมพิวเตชันและการลดความเสี่ยงของการทดลองจริงย่อมเพิ่มความน่าสนใจของสตาร์ทอัพไบโอไทยต่อผู้ลงทุนทั้งในและต่างประเทศ ข้อมูลแนวโน้มระดับภูมิภาคแสดงให้เห็นว่าเม็ดเงินลงทุนในสตาร์ทอัพเทคโนโลยีของเอเชียตะวันออกเฉียงใต้มีระดับประมาณ US$12–20 พันล้านต่อปี ในช่วงหลังปี 2020 โดยเฉพาะกลุ่มเฮลธ์เทคและไบโอมีอัตราการเติบโตเฉลี่ยราว 12–20% ต่อปี ซึ่งเมื่อผนวกกับผลลัพธ์เชิงต้นทุนและเวลา สตาร์ทอัพไทยที่มีเทคโนโลยีคัดกรองโมเลกุลเชิงคำนวณมีโอกาสดึงดูด VC เฉพาะทาง, กองทุน Corporate VC ของบริษัทเภสัชภัณฑ์, และพันธมิตรจากต่างประเทศ ทั้งในรูปแบบลงทุนโดยตรง, ข้อตกลงวิจัยร่วม, หรือสัญญาออกใบอนุญาต (licensing) ที่สร้างมูลค่าเพิ่มให้กับห่วงโซ่คุณค่า (value chain)
สรุปผลกระทบเชิงระบบที่สำคัญมีดังนี้
- เร่งเวลาเข้าสู่ตลาด (Faster go-to-market): ลดเวลาและจำนวนการทดลองจริง ช่วยให้ผลิตภัณฑ์นำร่องสามารถเข้าสู่รอบการทดสอบทางคลินิกหรือการใช้งานเชิงพาณิชย์ได้เร็วขึ้น
- กระตุ้นความร่วมมือข้ามศาสตร์: สร้างโมเดลความร่วมมือระหว่างนักวิจัย AI, นักเคมี และนักชีววิทยา ซึ่งช่วยยกระดับคุณภาพงานวิจัยและเพิ่มศักยภาพการต่อยอดเชิงพาณิชย์
- ดึงดูดการลงทุนและสร้างมูลค่าเพิ่ม: การลดความเสี่ยงเชิงวิทยาศาสตร์และการย่นเวลาพัฒนาผลิตภัณฑ์ทำให้น่าสนใจต่อกลุ่มนักลงทุนเฉพาะทางและพันธมิตรเชิงอุตสาหกรรม สร้างช่องทางรายได้ผ่านการร่วมวิจัยและการอนุญาตใช้เทคโนโลยี
- สร้างแรงงานทักษะสูง: เพิ่มความต้องการบุคลากรด้าน AI/ML, เคมีเชิงคำนวณ และชีวสารสนเทศ ช่วยยกระดับองค์ความรู้ภายในประเทศและลดการพึ่งพาทักษะจากต่างประเทศ
- ตั้งเงื่อนไขใหม่สำหรับนโยบายและโครงสร้างพื้นฐาน: จำเป็นต้องมีกรอบการจัดการข้อมูล การคุ้มครองทรัพย์สินทางปัญญา และการสนับสนุนจากภาครัฐในรูปแบบเงินทุนต้นแบบ (seed grants), tax incentives และคลัสเตอร์วิจัยร่วม
โดยรวมแล้ว การนำ Active Learning และ Foundation Models มาประยุกต์ใช้ในสตาร์ทอัพไบโอไทยเป็นตัวเร่งสำคัญที่จะเปลี่ยนโฉมภูมิทัศน์นวัตกรรมชีวภาพของประเทศ ทั้งในเชิงเศรษฐกิจและเชิงวิชาชีพ หากมีการออกแบบนโยบายสนับสนุน การส่งเสริมความร่วมมือเชิงระบบ และการลงทุนในคนและโครงสร้างพื้นฐานอย่างเหมาะสม ผลตอบแทนทั้งในรูปแบบงาน วิทยาศาสตร์ และมูลค่าทางเศรษฐกิจจะตามมาอย่างยั่งยืน
ข้อเสนอเชิงนโยบายและแนวทางปฏิบัติที่แนะนำ
ข้อเสนอเชิงนโยบายและแนวทางปฏิบัติที่แนะนำ
เพื่อส่งเสริมการนำ Active Learning และ Foundation Models มาใช้ในกระบวนการค้นคว้าสารตั้งต้นและคัดกรองโมเลกุลของสตาร์ทอัพไบโอไทยอย่างปลอดภัยและมีประสิทธิผล จำเป็นต้องออกแบบชุดมาตรการเชิงนโยบายที่ครอบคลุมทั้งด้านการสนับสนุนทางการเงิน โครงสร้างพื้นฐานข้อมูล มาตรฐานการแลกเปลี่ยนข้อมูล และการพัฒนากำลังคนข้ามศาสตร์ โดยมาตรการเหล่านี้ควรเน้นการสร้างระบบนิเวศที่เอื้อต่อความร่วมมือระหว่างภาครัฐ ภาคเอกชน และสถาบันการศึกษา เพื่อให้เทคโนโลยีใหม่สามารถทดสอบและปรับใช้ได้อย่างรวดเร็วแต่ยังคงความปลอดภัยต่อผู้ป่วยและความน่าเชื่อถือของข้อมูลวิจัย
ประการแรก ควรมีการจัดสรร ทุน R&D และโครงการนำร่องร่วม (public-private partnerships) ที่มุ่งเน้นการบูรณาการ Active Learning กับ Foundation Models ในผู้เล่นเชิงชีวเภสัชกรรมในประเทศ โดยมาตรการแนะนำได้แก่:
- กองทุนร่วมทุนเชิงยุทธศาสตร์ ระหว่างรัฐและเอกชนสำหรับโครงการนำร่อง 3–5 ปี เพื่อสนับสนุนการพัฒนาโมเดล การทำ validation ด้วยข้อมูลจริง และการเชื่อมต่อกับห้องปฏิบัติการต้นแบบ (pilot labs)
- แรงจูงใจทางการเงิน เช่น เครดิตภาษีแบบ R&D tax credit, matching grants และเงินอุดหนุนแบบ milestone-based เพื่อช่วยลดความเสี่ยงของสตาร์ทอัพเมื่อทดสอบเทคโนโลยีใหม่
- กรอบการประเมินผลร่วม ที่กำหนดตัวชี้วัดทางธุรกิจและวิทยาศาสตร์ (เช่น อัตราการค้นพบโมเลกุลที่เป็นไปได้ ลดระยะเวลานำไปทดสอบจริง) เพื่อให้การลงทุนมีความโปร่งใสและวัดผลได้
ประการที่สอง การสร้าง โครงสร้างพื้นฐานข้อมูลเชิงเปิดและมาตรฐานข้อมูล เป็นหัวใจสำคัญต่อการแชร์ข้อมูลอย่างปลอดภัยและการทำงานร่วมกันของโมเดลใหญ่ คำแนะนำเชิงปฏิบัติรวมถึง:
- จัดตั้งแพลตฟอร์มข้อมูลกลางที่สนับสนุนหลักการ FAIR (Findable, Accessible, Interoperable, Reusable) พร้อมนโยบายควบคุมการเข้าถึงและการคุ้มครองข้อมูลส่วนบุคคลตาม PDPA
- กำหนดมาตรฐานเมตาดาต้าและรูปแบบทางเคมีชีวภาพที่เป็นสากล (เช่น สนับสนุนการใช้ SMILES, InChI, และการแมปข้อมูลให้สอดคล้องกับฐานข้อมูลสากลอย่าง ChEMBL/ PubChem) เพื่อให้ Foundation Models สามารถใช้งานข้ามองค์กรได้โดยลดภาระการแปลงข้อมูล
- ส่งเสริมเทคนิคการแชร์ข้อมูลเช่น federated learning, differential privacy และการเข้ารหัสข้อมูล เพื่ออนุญาตให้โมเดลเรียนรู้จากข้อมูลกระจายโดยไม่เปิดเผยข้อมูลดิบ
- พัฒนากระบวนการรับรอง (certification) สำหรับชุดข้อมูลและผู้ให้บริการข้อมูล เพื่อยืนยันความถูกต้อง ปลอดภัย และความสามารถใช้งานร่วมกัน
ประการที่สาม การพัฒนากำลังคนข้ามศาสตร์และกระบวนการทดสอบในสภาพแวดล้อมควบคุมเป็นสิ่งจำเป็น ทั้งนี้ควรประกอบด้วย:
- โปรแกรมฝึกอบรมข้ามศาสตร์ ที่ออกแบบร่วมกันโดยมหาวิทยาลัย ภาคอุตสาหกรรม และหน่วยงานภาครัฐ ครอบคลุมความรู้ด้าน ML/AI, เคมีคอมพิวเตอร์, ชีววิทยาสังเคราะห์ และการกำกับดูแลความปลอดภัย เช่น หลักสูตรระยะสั้น หลักสูตรประกาศนียบัตร และทุนฝึกอบรม
- โครงการแลกเปลี่ยนและ secondment ให้ผู้เชี่ยวชาญด้าน AI ทำงานร่วมกับนักวิจัยในห้องปฏิบัติการ และในทางกลับกัน เพื่อเสริมสร้างความเข้าใจระหว่างศาสตร์
- การอบรมเจ้าหน้าที่กำกับดูแล (regulators) ให้มีความเข้าใจในเทคโนโลยี Active Learning และ Foundation Models เพื่อให้การประเมินความเสี่ยงและการอนุญาตเชิงนโยบายมีความรวดเร็วและมีมาตรฐาน
ประการที่สี่ เสนอให้จัดตั้ง regulatory sandbox เฉพาะด้านไบโอเทคและการค้นคว้ายา เพื่อให้สตาร์ทอัพและผู้พัฒนาสามารถทดสอบระบบในสภาพแวดล้อมที่มีกฎเกณฑ์ยืดหยุ่นภายใต้การกำกับดูแล โดยแนวทางปฏิบัติที่ควรนำไปใช้ได้แก่:
- กำหนดเกณฑ์ความปลอดภัยและขอบเขตการทดสอบชัดเจน เช่น ไม่อนุญาตการทดลองที่มีความเสี่ยงต่อมนุษย์โดยตรงในขั้นต้น และต้องมีแผนการป้องกันความเสี่ยงทางชีวภาพ
- ออกแบบกระบวนการอนุญาตแบบเป็นขั้นตอน (phased approvals) ที่เริ่มจากการจำลอง การทดสอบในห้องปฏิบัติการ และขยายสู่การทดลองร่วมกับภาคการแพทย์เมื่อผ่านเกณฑ์ชี้วัดความปลอดภัย
- สร้างกลไกการกำกับดูแลร่วม (joint oversight) ระหว่างหน่วยงานที่รับผิดชอบด้านยา สาธารณสุข และความปลอดภัยดิจิทัล เพื่อประเมินผลลัพธ์และบทเรียนเชิงนโยบายอย่างต่อเนื่อง
โดยสรุป มาตรการผสมผสานระหว่างทุนสนับสนุน โครงสร้างพื้นฐานข้อมูลมาตรฐาน การพัฒนากำลังคน และ sandbox ทางกฎระเบียบ จะช่วยให้สตาร์ทอัพไบโอไทยสามารถนำ Active Learning และ Foundation Models มาใช้อย่างมีประสิทธิภาพ ปลอดภัย และเพิ่มขีดความสามารถในการแข่งขันระหว่างประเทศ ผลลัพธ์ที่คาดหวังรวมถึงการลดต้นทุนการทดลองจริงและรอบเวลาในการค้นพบโมเลกุลนำร่อง ซึ่งจะเป็นปัจจัยสำคัญในการขับเคลื่อนเศรษฐกิจนวัตกรรมของประเทศในระยะกลางถึงยาว
บทสรุป
การผสานเทคนิค Active Learning เข้ากับ Foundation Models ช่วยให้สตาร์ทอัพไบโอไทยสามารถคัดกรองโมเลกุลนำร่องได้อย่างมีประสิทธิภาพมากขึ้น โดย Active Learning ช่วยเลือกตัวอย่างการทดลองที่ให้ข้อมูลมากที่สุด ขณะที่ Foundation Models ให้ความสามารถในการคาดการณ์และสังเคราะห์ความรู้จากข้อมูลขนาดใหญ่ร่วมกัน ผลลัพธ์คือการลดต้นทุนและจำนวนการทดลองในห้องปฏิบัติการจริงอย่างมีนัยสำคัญ ทำให้วงจรการทดลองสามารถทำซ้ำ-ปรับปรุงได้เร็วขึ้น เพิ่มโอกาสในการพัฒนาโมเลกุลที่มีศักยภาพเชิงพาณิชย์ และลดอุปสรรคทางการเงินสำหรับทีมงานสตาร์ทอัพที่มีทรัพยากรจำกัด
ความสำเร็จของแนวทางนี้ขึ้นกับปัจจัยพื้นฐานหลายประการ ได้แก่ ข้อมูลคุณภาพ และมาตรฐานการวัดผลที่สอดคล้องกัน ระบบแลกเปลี่ยนข้อมูลและการแต่งข้อมูล (data curation) ที่เข้มแข็ง ความร่วมมือระหว่างภาคการศึกษา อุตสาหกรรม และหน่วยงานกำกับดูแล รวมถึงนโยบายสนับสนุนที่ชัดเจนเพื่อสร้างแรงจูงใจและกรอบกติกาที่ปลอดภัยในระยะยาว ในด้านอนาคต หากมีการลงทุนในโครงสร้างพื้นฐานด้านข้อมูล การฝึกทักษะแรงงาน และกรอบกฎระเบียบที่เอื้อต่อการทดลองร่วมกัน เทคโนโลยีชุดนี้มีศักยภาพจะย่นระยะเวลาเข้าสู่ตลาด ลดต้นทุนการพัฒนายา ขยายไปสู่การแพทย์แม่นยำ และผลักดันให้ประเทศไทยเป็นศูนย์กลางนวัตกรรมไบโอในภูมิภาค โดยต้องอาศัยแผนปฏิบัติการร่วม (public–private partnerships) และมาตรการสนับสนุนที่ชัดเจนเพื่อให้เกิดระบบนิเวศที่ยั่งยืน

