เมื่อคิดถึงการค้นพบยาหรือการออกแบบโมเลกุลใหม่ ภาพจำแบบเดิมคือกระบวนการใช้เวลาหลายปีและงบประมาณหลายพันล้านบาท แต่ในยุคนี้ Machine Learning (ML) กำลังทลายกำแพงเหล่านั้น เปลี่ยนงานวิจัยเชิงทดลองที่ใช้เวลานานให้กลายเป็นกระบวนการที่สามารถจำลอง วิเคราะห์ และคาดการณ์ได้อย่างรวดเร็ว ด้วยโมเดลเช่น deep learning, graph neural networks และ generative models บริษัทสตาร์ทอัพและผู้บุกเบิกในอุตสาหกรรมยารายใหญ่รายงานการค้นพบโมเลกุลนำร่องและการคัดกรองสารตั้งต้นที่เร็วยิ่งขึ้น ขณะที่การลงทุนในเทคโนโลยี AI เพื่อการค้นพบยายังขยายตัวอย่างรวดเร็ว ส่งผลให้ความหวังในการลดต้นทุนและเพิ่มอัตราความสำเร็จของการพัฒนายาเป็นรูปธรรมมากขึ้น
บทความนี้จะพาคุณสำรวจภาพรวมตั้งแต่เทคโนโลยีหลักและกระบวนการปฏิบัติจริง (เช่น virtual screening, de‑novo design, การทำนาย ADMET และการหาโอกาสทำ drug repurposing) ไปจนถึงกรณีศึกษาจริงที่แสดงผลลัพธ์เชิงปฏิบัติ พร้อมวิเคราะห์ข้อจำกัดด้านข้อมูล ความโปร่งใสของโมเดล กรอบกฎระเบียบ และความท้าทายในเชิงการนำไปใช้จริง นอกจากนี้เราจะมองไปยังทิศทางอนาคตที่เป็นไปได้ เช่นการรวม ML กับหุ่นยนต์ในห้องปฏิบัติการ ข้อมูลองค์ความรู้แบบหลายมิติ (multi‑omics) และการปรับการรักษาให้เหมาะกับบุคคล (precision medicine) เพื่อวาดภาพว่าการค้นพบยาโดยใช้ Machine Learning จะเปลี่ยนแปลงอนาคตวงการแพทย์อย่างไร
บทนำ: ทำไม Machine Learning จึงสำคัญต่อการค้นพบยา
บทนำ: ทำไม Machine Learning จึงสำคัญต่อการค้นพบยา
การค้นพบและพัฒนายาแบบดั้งเดิมเป็นกระบวนการที่ใช้เวลายาวนานและมีความเสี่ยงสูง โดยทั่วไปการเดินทางจากการค้นพบโมเลกุลเริ่มต้นจนถึงการได้รับอนุมัติใช้รักษาในมนุษย์ใช้เวลาประมาณ 10–15 ปี และมีต้นทุนรวมในระดับ ร้อยล้านถึงพันล้านดอลลาร์สหรัฐต่อโมเลกุล การลงทุนมหาศาลนี้ยังมาพร้อมกับอัตราความล้มเหลวสูง — มีเพียงส่วนน้อยของโมเลกุลที่ผ่านด่านก่อนคลินิกและการทดลองทางคลินิกจนได้รับอนุมัติจริง ทำให้บริษัทเภสัชกรรมและนักลงทุนต้องแบกรับความเสี่ยงด้านเวลและต้นทุนอย่างหนัก
ภายใต้บริบทดังกล่าว Machine Learning (ML) ได้กลายเป็นเทคโนโลยีสำคัญที่หลายองค์กรมองว่าเป็นเครื่องมือในการพลิกโฉมกระบวนการค้นพบยา ML ช่วยวิเคราะห์ข้อมูลขนาดใหญ่จากแหล่งต่าง ๆ เช่น ข้อมูลจีโนม โปรตีน โครงสร้างโมเลกุล ข้อมูลทางคลินิก และข้อมูลการทดลองในห้องปฏิบัติการ เพื่อค้นหาแพทเทิร์นที่มนุษย์อาจมองไม่เห็นและเร่งการตัดสินใจเชิงวิทยาศาสตร์
บทบาทของ ML ถูกนำไปใช้ในหลายขั้นตอนสำคัญของการค้นพบยา เช่น:
- Target identification — คัดกรองและระบุเป้าหมายทางชีวภาพที่มีความน่าจะเป็นสูงต่อการเป็นจุดยับยั้งหรือกระตุ้นเชิงรักษา
- Virtual screening — ประเมินและคัดกรองสารนับล้านถึงพันล้านโมเลกุลเชิงคอมพิวเตอร์ เพื่อลดจำนวนสารที่ต้องทดสอบในห้องปฏิบัติการ
- Lead optimization — ปรับปรุงคุณสมบัติของโมเลกุลนำโดยการทำนายความสัมพันธ์โครงสร้าง-กิจกรรม (SAR) อย่างรวดเร็ว
- การพยากรณ์ ADMET (absorption, distribution, metabolism, excretion, toxicity) — ลดความเสี่ยงด้านความเป็นพิษและปัญหาฟาร์มาโคคิเนติกก่อนเข้าสู่การทดลองคลินิก
จากมุมมองเชิงเศรษฐกิจและการลงทุน แนวโน้มชัดเจนว่าอุตสาหกรรมให้ความสำคัญกับ ML มากขึ้น โดยในทศวรรษที่ผ่านมาเงินลงทุนในสตาร์ทอัพและโครงการที่มุ่งใช้ AI/ML ในการค้นพบยาเพิ่มขึ้นอย่างรวดเร็ว การระดมทุนระดับพันล้านดอลลาร์ในระดับสะสมและการเติบโตของการลงทุนรายปีสะท้อนความคาดหวังว่าการนำ ML มาใช้สามารถลดเวลาและต้นทุนในกระบวนการพัฒนายาได้อย่างมีนัยสำคัญ อีกทั้งยังเปิดโอกาสใหม่ ๆ เช่น การค้นพบยาเฉพาะกลุ่มผู้ป่วย (precision medicine) และการรีพอร์ปสิงค์ยาเดิม (drug repurposing) ซึ่งทั้งหมดนี้ช่วยเพิ่มประสิทธิภาพการลงทุนและลดความเสี่ยงในพอร์ตโฟลิโอการพัฒนายาของบริษัท
กระบวนการค้นพบยา: จากรูปแบบดั้งเดิมสู่ ML-driven pipeline
การค้นพบยาแบบดั้งเดิมเป็นกระบวนการเชิงลำดับที่เริ่มจากการระบุเป้าหมาย (target identification) ตามด้วยการคัดกรองสารค้นหาผู้ทำปฏิกิริยา (hit discovery), การพัฒนา hit ให้เป็น lead (hit-to-lead), การเพิ่มประสิทธิภาพ (lead optimization) และการประเมินคุณสมบัติทางเภสัชจลนศาสตร์และความปลอดภัย (ADMET) ก่อนเข้าสู่การทดลองก่อนคลินิกและคลินิก กระบวนการนี้มักใช้เวลาเป็นปีถึงทศวรรษ และมีจุดบกพร่องสำคัญเช่น ความจำกัดด้านปริมาณข้อมูลเชิงทดลอง ความเบี่ยงเบนของการคัดเลือกสารจากการทดสอบในห้องปฏิบัติการ และต้นทุนในการสังเคราะห์และทดสอบตัวอย่างจำนวนมาก
การผนวก Machine Learning (ML) เข้ากับ pipeline ของการค้นพบยาได้เปลี่ยนแปลงรูปแบบการทำงานในหลายขั้นตอน โดย เร่งการตัดสินใจ, ลดปริมาณการทดลองที่ต้องทำจริง และเพิ่มโอกาสในการค้นพบสารที่มีศักยภาพสูงสุด ML ช่วยให้สามารถประมวลผลข้อมูลเชิงโครงสร้างชีวโมเลกุล ข้อมูลการทดลองเชิงชีวภาพ และข้อมูลจีโนมิกได้อย่างรวดเร็ว เพื่อสนับสนุนการตัดสินใจเชิงกลยุทธ์และการคัดเลือกเป้าหมายที่เหมาะสม

ขั้นตอนหลักของการค้นพบยาและจุดบกพร่องที่ ML สามารถแก้ไขได้
- Target identification: แบบเดิมพึ่งพาการค้นพบผ่านงานชีวภาพเชิงทดลองและการสืบค้นวรรณกรรม ซึ่งใช้เวลานานและมีความเสี่ยง ML สามารถวิเคราะห์ข้อมูลหลายมิติ (omics, ปฏิกิริยาทางชีวภาพ, โครงสร้างโปรตีน) เพื่อคาดการณ์เป้าหมายที่มีความเชื่อมโยงกับพยาธิสภาพ เพิ่มประสิทธิภาพการระบุ target ใหม่และลด false positives
- Virtual screening / Hit discovery: การสกรีนด้วยแล็บแบบเดิมต้องทดสอบสารเป็นจำนวนมาก ซึ่งมีต้นทุนสูง ML และ docking แบบเร่งความเร็วช่วยให้คัดกรองสารจากคลังโมเลกุลขนาดล้านถึงพันล้านรายการภายในเวลาเป็นวันหรือสัปดาห์ แทนที่จะต้องสังเคราะห์และทดสอบจริงหลายหมื่นตัวอย่าง การสกรีนเชิงคอมพิวเตอร์จึงลดค่าใช้จ่ายและเวลาได้อย่างมาก
- Hit-to-lead และ Lead optimization: ขั้นตอนดั้งเดิมมักอาศัยวงจรทดลอง-ออกแบบ-ทดสอบซ้ำหลายรอบ หลายปี ML (เช่น โมเดลการพยากรณ์ activity, กำลังการเรียนรู้เชิงเพิ่มค่า และ Bayesian optimization) ช่วยทำนายความสัมพันธ์โครงสร้าง-กิจกรรมและคุณสมบัติ ADMET ทำให้สามารถจำกัดชุดโมเลกุลที่จะสังเคราะห์จริงให้เหลือน้อยลง ตัวอย่างเชิงประจักษ์ชี้ว่า pipeline ที่ผนวก ML สามารถลดเวลา hit-to-lead จากระดับปีเหลือเป็นเดือนหรือไตรมาสในหลายกรณี
- ADMET prediction: การทดสอบ ADMET แบบ wet lab มีทั้งต้นทุนและเวลา ML และเทคนิคการเรียนรู้เชิงลึกช่วยพยากรณ์การดูดซึม เมตาบอไลต์ ความเป็นพิษ และการกระจายในร่างกายได้เร็วขึ้น ช่วยคัดกรองโมเลกุลที่มีโอกาสล้มเหลวทางคลินิกตั้งแต่เนิ่นๆ
ตัวอย่างผลประหยัดเวลาและต้นทุนในการนำ ML มาใช้
ในทางปฏิบัติ หลักเกณฑ์ที่เห็นได้ชัดคือการย่อตัวเลขรอบเวลาของการพัฒนา: การสกรีนเชิงคอมพิวเตอร์สามารถลดจำนวนโมเลกุลที่ต้องสังเคราะห์จริงลงเหลือเพียง หน่วยหรือหลักสิบเปอร์เซ็นต์ ของชุดเริ่มต้น ส่งผลให้ต้นทุนการทดลองด้านเคมีและชีวภาพลดลงอย่างมีนัยสำคัญ นอกจากนี้หลายองค์กรรายงานกรณีศึกษาที่การใช้ ML ในขั้นตอน hit-to-lead ช่วยลดระยะเวลาจากหลายปีเหลือเป็นเดือนถึงไตรมาส ในขณะที่อัตราขาดทุนของโครงการก่อนคลินิกก็ลดลงจากการคัดกรอง ADMET ที่มีประสิทธิภาพขึ้น
บทบาทของการผสมผสานข้อมูล in silico กับการทดลองแบบ wet lab
ประสิทธิผลสูงสุดของ ML เกิดขึ้นเมื่อมีการผสานข้อมูล in silico กับวงจรทดลองแบบ wet lab อย่างเป็นระบบ รูปแบบปฏิบัติที่ได้ผลดีคือการตั้งวงจร active learning ซึ่งโมเดล ML จะคาดการณ์ตัวอย่างที่มีคุณค่าสูงสุดสำหรับการสังเคราะห์และการทดสอบจริง ข้อมูลผลลัพธ์จากห้องปฏิบัติการจะถูกป้อนกลับมาปรับปรุงโมเดล ทำให้การเลือกตัวอย่างในรอบถัดไปแม่นยำขึ้น วงจรเช่นนี้ช่วยลดจำนวนการทดลองที่ไม่จำเป็นและเพิ่มอัตราสำเร็จของการค้นพบสารนำไปสู่การพัฒนาทางคลินิก
สรุปได้ว่า ML ไม่ได้มาทดแทนการทดลองในห้องปฏิบัติการทั้งหมด แต่เป็นเครื่องมือที่เพิ่มประสิทธิภาพการตัดสินใจและลดความเสี่ยงระหว่างทาง การผสมผสานอย่างสมดุลระหว่างการคาดการณ์เชิงคอมพิวเตอร์กับการยืนยันเชิงทดลองคือกุญแจสำคัญที่จะเร่งเวลา ลดต้นทุน และเพิ่มอัตราความสำเร็จในการค้นพบยาในอนาคต
เทคโนโลยีและโมเดลสำคัญที่ขับเคลื่อนการค้นพบยา
เทคโนโลยีและโมเดลสำคัญที่ขับเคลื่อนการค้นพบยา
การค้นพบยาในยุคปัจจุบันขับเคลื่อนด้วยชุดเทคโนโลยีด้าน deep learning ที่หลากหลาย โดยเฉพาะเมื่อต้องจัดการกับข้อมูลเชิงโครงสร้างของโมเลกุลและโปรตีน โมเดลเชิงกราฟ (Graph Neural Networks, GNNs) ถูกนำมาใช้อย่างแพร่หลายเพราะสามารถแทนโครงสร้างโมเลกุลเป็นกราฟได้อย่างเป็นธรรมชาติ—อะตอมเป็นโหนดและพันธะเป็นเส้นเชื่อม—ทำให้การเรียนรู้แบบเชิงโครงสร้างเพื่อพยากรณ์สมบัติทางเคมี เช่น ความเป็นพิษ ความสามารถในการจับกับเป้าหมาย และความสามารถในการละลาย เป็นไปได้ด้วยประสิทธิภาพสูง งานวิจัยหลายชิ้นในชุดข้อมูลมาตรฐานอย่าง MoleculeNet แสดงให้เห็นว่า GNNs มักให้ผลลัพธ์ที่ดีกว่ารูปแบบฟีเจอร์เชิงสถิติดั้งเดิม โดยเฉพาะเมื่อมีการเพิ่มฟีเจอร์เชิงทิศทางและข้อมูลเชิงอิเล็กทรอนิกส์เข้าไป
นอกจาก GNNs แล้ว generative models กลายเป็นเครื่องมือสำคัญสำหรับการออกแบบโมเลกุลใหม่ รูปแบบยอดนิยมได้แก่ Variational Autoencoders (VAEs), Generative Adversarial Networks (GANs) และกลุ่มของ diffusion models ซึ่งแต่ละแบบมีข้อได้เปรียบที่แตกต่างกัน เช่น VAEs สามารถเรียนรู้การแจกแจงเชิงต่อเนื่องของโมเลกุล ทำให้สะดวกต่อการสำรวจสภาพแวดล้อมเคมี ส่วน GANs ให้ความสามารถในการสร้างตัวอย่างที่สมจริง ในขณะที่ diffusion models (หรือ score-based models) แสดงความสามารถในการผลิตโมเลกุลที่มีความหลากหลายและมีความสอดคล้องทางเคมีสูง ตัวอย่างแอปพลิเคชันเชิงธุรกิจคือการใช้โมเดลเหล่านี้เพื่อออกแบบโมเลกุลที่คาดหวังจะมีคุณสมบัติทางเภสัช เช่น ความจำเพาะต่อเป้าหมาย ความคงตัวในร่างกาย และโปรไฟล์ความเป็นพิษต่ำ โดยโมเดลสามารถผสานเงื่อนไข (conditional generation) เพื่อเร่งการออกแบบไปยังพื้นที่เคมีที่ต้องการ
อีกเสาหลักที่เปลี่ยนเกมสำหรับการออกแบบยาคือโซลูชันพยากรณ์โครงสร้างโปรตีน เช่น AlphaFold จาก DeepMind และตามมาด้วยระบบอื่น ๆ เช่น RoseTTAFold ซึ่งช่วยให้สามารถคาดการณ์โครงสร้างสามมิติของโปรตีนได้ในระดับความละเอียดสูง จากการที่ระบบเหล่านี้เปิดเผยพยากรณ์ของโปรตีนจำนวนมากในฐานข้อมูลสาธารณะ ทำให้นักวิจัยสามารถเข้าถึงข้อมูลเชิงโครงสร้างของเป้าหมายทางชีวภาพได้อย่างรวดเร็วและครอบคลุม การมีโครงสร้างโปรตีนเชิงทฤษฎีที่เชื่อถือได้ช่วยให้กระบวนการ target-based design เช่น การคัดเลือกบริเวณการจับ (binding site) และการจำลองการจับคู่แบบโมเลกุล-โปรตีน (docking) มีความแม่นยำและมีประสิทธิภาพมากขึ้น ส่งผลให้วงจรการค้นพบยาสั้นลงและลดต้นทุนการทดลองลงอย่างมีนัยสำคัญ
การเรียนรู้แบบ self-supervised และ transfer learning เป็นเทคนิคที่เสริมความสามารถของโมเดล AI ในการค้นพบยาอย่างเป็นรูปธรรม โดยโมเดลถูกฝึกจากข้อมูลขนาดใหญ่ที่ไม่ได้ติดป้ายกำกับ (เช่น คอลเลกชันของ SMILES หรือกราฟโมเลกุลหลายล้านชิ้น) ผ่านงานที่ไม่ต้องการฉลาก เช่น การทำนายส่วนที่ถูกปิดบัง (masking) หรือการทำ contrastive learning หลังจากนั้นโมเดลที่ได้จะถูกปรับแต่ง (fine-tuned) กับชุดข้อมูลเฉพาะงานเช่น การทดสอบความเป็นพิษหรือกิจกรรมทางชีวภาพ กระบวนการนี้ช่วยลดความจำเป็นของข้อมูลติดป้ายจำนวนมาก และมักให้การทั่วไปที่ดีขึ้นเมื่อต้องย้ายไปยังงานใหม่ ๆ ตัวอย่างเช่น transformer-based models ที่เรียนรู้จาก SMILES/SELFIES หรือ GNN ที่ผ่านการ pretraining สามารถเพิ่มความแม่นยำในการพยากรณ์สมบัติเมื่อเทียบกับการฝึกจากศูนย์
สรุปประเด็นสำคัญในเชิงปฏิบัติ:
- GNNs เหมาะสมกับการแทนและเรียนรู้เชิงโครงสร้างของโมเลกุล ช่วยพยากรณ์สมบัติและฟังก์ชันทางเภสัชได้อย่างตรงจุด
- Generative models (VAEs, GANs, diffusion) เปิดทางสู่การออกแบบโมเลกุลใหม่ที่สอดคล้องกับเงื่อนไขเชิงเภสัชวิทยา ลดเวลาการค้นหาเคมีที่เหมาะสม
- โซลูชันพยากรณ์โครงสร้างโปรตีน เช่น AlphaFold ทำให้ target-based design เป็นไปได้รวดเร็วและแม่นยำขึ้น ลดการพึ่งพาการทดลองโครงสร้างที่มีราคาแพง
- Self-supervised และ transfer learning ช่วยให้โมเดลใช้ประโยชน์จากข้อมูลขนาดใหญ่ที่ไม่มีป้าย และย้ายความรู้ไปยังงานเฉพาะได้อย่างมีประสิทธิภาพ
การผสานเทคโนโลยีเหล่านี้เข้ากับกระบวนการค้นพบยา—ตั้งแต่การคัดเลือกเป้าหมาย การออกแบบโมเลกุล ไปจนถึงการประเมินความเป็นไปได้เชิงคลินิก—เป็นปัจจัยสำคัญที่เปลี่ยนสนามแข่งขันในอุตสาหกรรมยา ทั้งในแง่ของความเร็ว ต้นทุน และโอกาสในการค้นพบโมเลกุลที่มีศักยภาพสูงสำหรับการทดลองระยะต้นและการนำไปสู่การพัฒนาทางคลินิก
กรณีศึกษา: ความสำเร็จและบทเรียนจากบริษัทและโครงการชั้นนำ
กรณีศึกษา: ความสำเร็จและบทเรียนจากบริษัทและโครงการชั้นนำ
AlphaFold ของ DeepMind เป็นหนึ่งในความสำเร็จที่จับต้องได้ชัดเจนที่สุดในเชิงวิทยาศาสตร์ของการประยุกต์ใช้ Machine Learning ด้านชีวภาพ ในปี 2020 AlphaFold ทำผลงานโดดเด่นในการแข่งขัน CASP14 และต่อมาในปี 2021 มีการเปิดตัว AlphaFold DB ซึ่งประกอบด้วยผลคาดการณ์โครงสร้างโปรตีนมากกว่า 350,000 โครงสร้าง ครอบคลุมโปรตีนของสิ่งมีชีวิตหลากหลายสายพันธุ์รวมถึงเกือบทั้งชุดโปรตีนมนุษย์ (~98.5%) การเข้าถึงข้อมูลโครงสร้างในวงกว้างนี้ได้เปลี่ยนจากการรอผลทดลอง X‑ray หรือ cryo‑EM ที่ใช้เวลาหลายเดือนหรือหลายปี มาสู่การมีข้อมูลคาดการณ์ที่ช่วยเร่งการออกแบบแบบ structure-based drug design (SBDD) ทำให้ทีมนักวิจัยสามารถระบุไซต์ยึดจับ สำรวจรูปร่างโมเลกุล และเริ่มทำการคัดเลือกสารนำทดลองได้เร็วขึ้นอย่างมีนัยสำคัญ

Exscientia และ Insilico Medicine เป็นตัวอย่างบริษัทสตาร์ทอัพที่นำกระบวนการ AI เข้ามาใช้ในการออกแบบโมเลกุลและผลักดันเข้าสู่การทดลองทางคลินิก โดย Exscientia ประกาศความสำเร็จของ DSP‑1181 ซึ่งถูกระบุว่าเป็นหนึ่งในโมเลกุลที่ออกแบบด้วย AI รุ่นแรก ๆ ที่เข้าสู่การทดลองระยะต้น (Phase I) ในปี 2020 ขณะที่ Insilico ได้เผยแพร่กรณีศึกษาที่ระบุว่าระบบของตนสามารถออกแบบตัวนำทดลองที่มีคุณสมบัติเป้าหมายได้ภายในระยะเวลาสั้นเมื่อเทียบกับกระบวนการแบบดั้งเดิม ทั้งสองบริษัทรายงานว่า AI ช่วยลดวงจรการค้นพบโมเลกุลนำทดลองจากกรอบเวลาหลายปีเหลือเป็นเดือนหรือปีเดียวในขั้นต้น ซึ่งสะท้อนประสิทธิภาพด้านความเร็วและการคัดกรองเชิงคำนวณที่สูงขึ้น
BenevolentAI เป็นตัวอย่างกรณีการนำ AI มาใช้ในเชิงรีพพอสซิ่ง (drug repurposing) ในช่วงการระบาดของ COVID‑19 บริษัทใช้กราฟความรู้ (knowledge graph) และเทคนิคการประมวลผลภาษาเพื่อค้นหาโมเลกุลที่มีศักยภาพในการยับยั้งเส้นทางการติดเชื้อและการอักเสบ และเสนอสารที่ต่อมาถูกนำไปพิจารณาในการทดลองทางคลินิกต่อไป แม้ว่าการตอบรับทางคลินิกจะไม่ใช่การยืนยันผลแน่ชัดในทุกกรณี แต่กรณีของ BenevolentAI แสดงให้เห็นศักยภาพของ AI ในการสร้างไอเดียรีพพอสซิ่งที่รวดเร็วและขยายขอบเขตการสำรวจยาที่มนุษย์อาจละเลย
บทวิเคราะห์: ผลลัพธ์ จุดแข็ง และข้อจำกัด
- จุดแข็ง — AI ช่วยเร่งกระบวนการคัดกรองและออกแบบโมเลกุล ลดเวลาและต้นทุนในขั้นตอนค้นพบ สามารถวิเคราะห์ข้อมูลปริมาณมากและเชื่อมโยงข้อมูลเชิงลึกจากหลายแหล่งได้ เช่น การใช้โครงสร้างจาก AlphaFold เพื่อขับเคลื่อน SBDD และการใช้โมเดลการออกแบบเพื่อผลิตโมเลกุลนำทดลองที่มีลักษณะตามข้อกำหนด
- ผลลัพธ์ที่จับต้องได้ — ตัวอย่างเช่น DSP‑1181 (Exscientia) ที่เข้าสู่การทดลองในมนุษย์ และผลงานการคัดเลือกสารรีพพอสซิ่งของ BenevolentAI ที่ถูกนำไปสู่การศึกษาเพิ่มเติม แสดงให้เห็นการแปลผลงานเชิงคอมพิวเตอร์ไปสู่ขั้นตอนการทดสอบจริงได้
- ข้อจำกัด — แม้ AI จะให้การคาดการณ์ที่รวดเร็ว แต่ความแม่นยำเชิงชีวภาพและการพยากรณ์คุณสมบัติ ADMET (การดูดซึม การกระจาย การเมแทบอลิซึม การขับถ่าย และความเป็นพิษ) ยังมีความท้าทายใหญ่ ข้อมูลฝึกสอนมีอคติหรือขาดคุณภาพจะนำไปสู่ผลลวง และโมเดลไม่สามารถทดแทนการทดลองทางชีวภาพและการทดสอบทางคลินิกที่จำเป็นสำหรับการยืนยันความปลอดภัยและประสิทธิผล
บทเรียนสำคัญ ที่องค์กรและนักลงทุนควรนำไปใช้คือความจำเป็นของการผนวกข้อมูลทดลองจริงเข้ากับพลังของ AI—ไม่ว่าจะเป็นข้อมูลการทดลอง in vitro, in vivo, ข้อมูลโครงสร้างจากเทคนิคเชิงทดลอง หรือข้อมูลคลินิก การทำงานแบบวงจรป้อนกลับ (iterative loop) ระหว่างห้องปฏิบัติการและทีม AI เป็นสิ่งจำเป็นเพื่อปรับปรุงโมเดลอย่างต่อเนื่อง นอกจากนี้การมาตรฐานข้อมูล การตรวจสอบผลแบบเบี่ยงเบน (bias auditing) และการร่วมมือระหว่างนักชีววิทยา ผู้เชี่ยวชาญเภสัชวิทยา และนักวิทยาศาสตร์ข้อมูลจะช่วยเร่งการแปลผลจากคอมพิวเตอร์สู่ผู้ป่วยได้อย่างมีความรับผิดชอบ
สรุป: กรณีของ AlphaFold, Exscientia, Insilico และ BenevolentAI แสดงให้เห็นว่าการประยุกต์ใช้ Machine Learning สามารถเปลี่ยนโมเดลการค้นพบยาให้เร็วขึ้นและขยายขอบเขตการค้นหาได้จริง แต่อย่างไรก็ตาม ความสำเร็จเชิงคอมพิวเตอร์ต้องมาพร้อมกับการตรวจสอบเชิงชีวภาพที่เข้มงวดและการบูรณาการข้อมูลทดลองเพื่อให้เกิดการแปลผลที่ปลอดภัยและมีประสิทธิผลในเชิงคลินิก
ข้อมูล แหล่งข้อมูล และการจัดการข้อมูลเป็นหัวใจของความแม่นยำ
ข้อมูล แหล่งข้อมูล และการจัดการข้อมูลเป็นหัวใจของความแม่นยำ
การค้นพบยาใหม่ด้วย Machine Learning ขึ้นอยู่กับ คุณภาพของข้อมูล เป็นสำคัญ — ไม่ว่าจะเป็นผลการทดลองเชิงชีวภาพ (high-quality assays), ข้อมูลโครงสร้างโปรตีน (structural data), ข้อมูลเชิงโมเลกุลจากโอมิกส์ (omics) หรือข้อมูลจากสภาพคลินิกจริง (real-world data) เช่น EHRs ข้อมูลที่ชัดเจน ถูกต้อง และมีเมตาดาต้าครบถ้วน จะช่วยให้โมเดลเรียนรู้รูปแบบทางชีววิทยาที่แท้จริงและลดความเสี่ยงของการคาดการณ์ที่ผิดพลาด ตัวอย่างเช่น ฐานข้อมูลโครงสร้างโปรตีน PDB มีข้อมูลโครงสร้างระดับอะตอมหลายแสนชิ้นที่เป็นรากฐานในการทำ structure-based drug design ขณะที่ AlphaFold DB ได้ขยายขอบเขตด้วยการคาดการณ์โครงสร้างมากกว่า 200 ล้านโครงสร้าง ซึ่งช่วยเติมเต็มช่องว่างของข้อมูลทางโครงสร้างในการพัฒนายา
แหล่งข้อมูลสาธารณะที่นิยมใช้ในการพัฒนายาและโมเดล ML ได้แก่:
- PDB (Protein Data Bank) — แหล่งข้อมูลโครงสร้างโปรตีนเชิงทดลองและการคาดการณ์
- ChEMBL — ฐานข้อมูลกิจกรรมทางเภสัชวิทยาของสารเคมี มีข้อมูลของสารตั้งแต่ล้านถึงหลายล้านรายการ
- PubChem — ฐานข้อมูลสารเคมีสาธารณะขนาดใหญ่ (มากกว่า 100 ล้านสาร)
- UniProt — ฐานข้อมูลลำดับโปรตีนและการคัดแยกหน้าที่ของโปรตีน
- OMICS repositories (เช่น GEO, ArrayExpress, ENA) — ข้อมูลระดับจีโนม, ทรานสคริปโตม, โปรตีโอมิกส์ จากหลายโครงการและตัวอย่างเป็นล้านชิ้น
- EHRs และ Real-World Data — ข้อมูลผู้ป่วยจริงจากระบบสุขภาพและฐานข้อมูลการรักษา ซึ่งมีบทบาทในการประเมินประสิทธิผลและความปลอดภัยของยาในโลกความเป็นจริง
- clinicaltrials.gov — บันทึกการทดลองทางคลินิกเชิงระบบที่ช่วยในการติดตามผลการทดลองและการรายงานผลลัพธ์
แม้จะมีแหล่งข้อมูลมากมาย แต่ปัญหาคุณภาพข้อมูลยังคงเป็นอุปสรรคใหญ่ ได้แก่ batch effects (ความแตกต่างที่เกิดจากชุดการทดลองหรือผู้ปฏิบัติการต่างกัน), measurement noise (ความผิดพลาดในการวัด), และ label bias (ป้ายกำกับหรือฉลากที่มีความลำเอียง) ปัญหาเหล่านี้สามารถนำไปสู่โมเดลที่มีประสิทธิภาพลดลงหรือให้ผลลวง ตัวอย่างเช่น การรวมข้อมูล omics จากหลายห้องปฏิบัติการโดยไม่แก้ไข batch effects อาจทำให้สัญญาณชีวภาพที่แท้จริงถูกบดบังและสร้าง false positives ที่สูงขึ้น สำหรับข้อมูลจาก EHRs ยังมีปัญหาเช่นค่าที่หายไป (missingness), การบันทึกที่ไม่สอดคล้อง และความไม่เป็นมาตรฐานของรหัสวินิจฉัย (ICD, SNOMED) ซึ่งต้องการการจัดการเชิงระบบ
แนวทางแก้ไขเชิงปฏิบัติและกลยุทธ์ทางเทคนิคที่สำคัญ ได้แก่:
- Data curation และ standardization: ตรวจสอบแหล่งที่มา, ทำ normalization, ใช้ ontology และมาตรฐานเมตาดาต้า (FAIR principles) เพื่อเพิ่มความสามารถในการทำซ้ำและการบูรณาการข้อมูล
- Batch-effect correction และ noise reduction: ใช้วิธีการเช่น ComBat, quantile normalization หรือวิธีการสถิติอื่นๆ เพื่อลดความเบี่ยงเบนข้ามชุดข้อมูล
- การจัดการความไม่สมดุลของข้อมูล: ใช้การ oversampling, undersampling, class weighting, สูตรค่าเสียหายแบบ focal loss หรือสร้างตัวอย่างสังเคราะห์ด้วย SMILES augmentation หรือ graph augmentation ในเคมิคอลส์
- Data augmentation และการสร้างข้อมูลสังเคราะห์: ใช้ generative models (GANs, VAE) สำหรับสร้างตัวอย่างโมเลกุลหรือสัญญาณ omics เสริม เพื่อเพิ่มตัวอย่างในคลาสที่มีน้อย
- Federated learning: เมื่อต้องทำงานกับข้อมูลที่ไม่สามารถถ่ายโอนข้ามสถาบันได้ (เช่น EHRs) federated learning ช่วยให้ฝึกโมเดลร่วมกันได้โดยไม่รวมข้อมูลดิบ ลดความเสี่ยงด้านความเป็นส่วนตัวและปัญหาทางกฎหมาย
- Transfer learning: การนำโมเดลที่ได้รับการฝึกจากโดเมนหนึ่งมาปรับใช้ในโดเมนอื่น (เช่น ใช้โมเดลที่เรียนรู้จากชุดข้อมูลเคมีขนาดใหญ่แล้ว fine-tune กับข้อมูลเฉพาะของโรค) ช่วยลดความต้องการข้อมูลจำนวนมากและเร่งเวลาในการพัฒนา
- Governance และ data provenance: ติดตามการเปลี่ยนแปลงข้อมูลด้วย data versioning, ระบุแหล่งที่มาและกระบวนการ preprocessing เพื่อสร้างความเชื่อมั่นทางธุรกิจและการตรวจสอบย้อนหลัง
ในเชิงธุรกิจ การลงทุนในกระบวนการจัดการข้อมูล (data pipelines, curation teams, metadata standards) และเทคโนโลยีปกป้องข้อมูล (เช่น federated learning, differential privacy) เป็นการลงทุนที่คุ้มค่า เพราะช่วยลดความเสี่ยงของการตัดสินใจบนพื้นฐานข้อมูลที่บิดเบี้ยวและเพิ่มความแม่นยำของโมเดล นอกจากนี้ การใช้ transfer learning และการผสมผสานแหล่งข้อมูลสาธารณะ เช่น PDB, ChEMBL และ PubChem กับข้อมูลภายในองค์กร จะช่วยเร่งการค้นพบยาใหม่และลดต้นทุนการวิจัยในระยะยาว
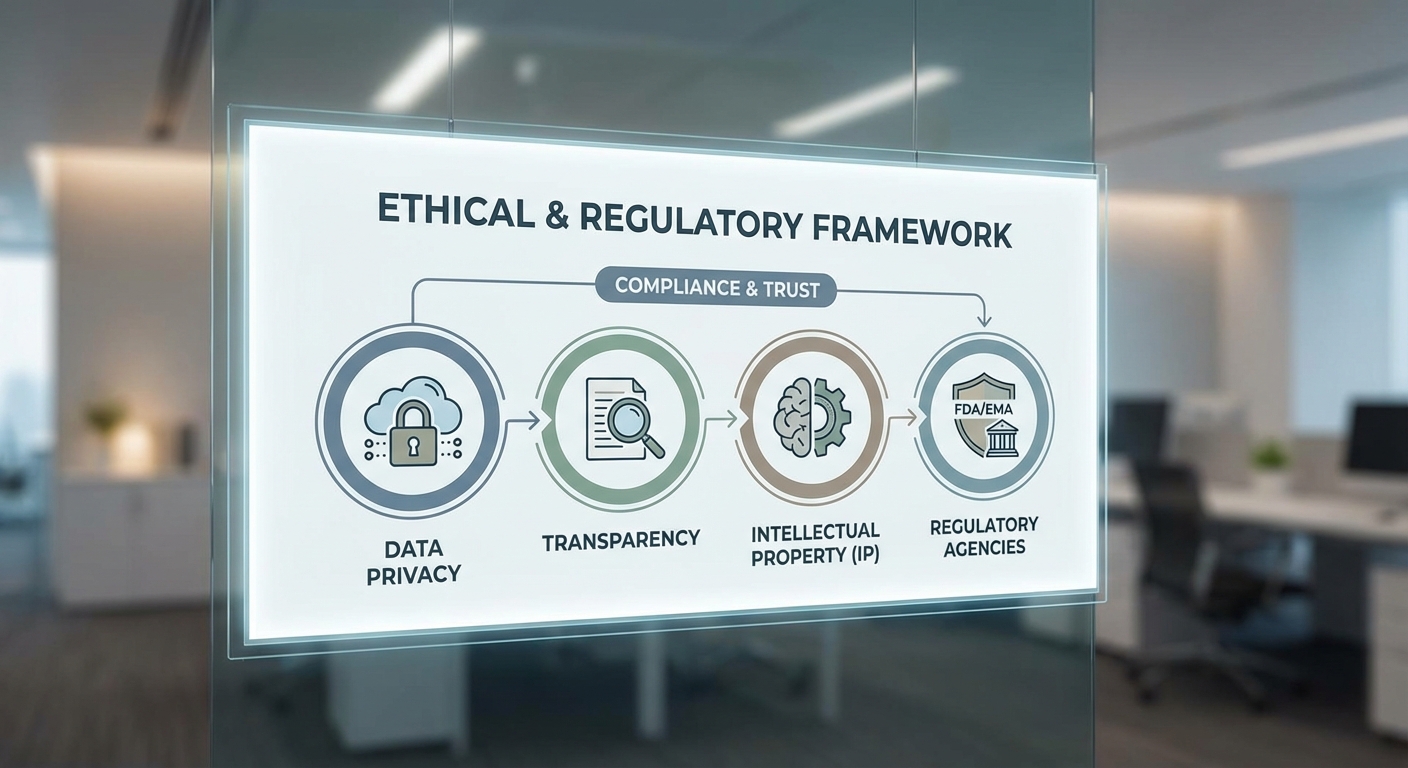
ความท้าทายด้านจริยธรรม กฎหมาย และการกำกับดูแล
ความท้าทายด้านจริยธรรม กฎหมาย และการกำกับดูแล
การนำ Machine Learning มาใช้ในการค้นพบยาใหม่ให้ประโยชน์มหาศาล แต่ก็สร้างความท้าทายด้านจริยธรรม กฎหมาย และการกำกับดูแลที่ต้องได้รับการจัดการอย่างรัดกุมก่อนนำไปสู่การทดลองทางคลินิกหรือการใช้ในระบบสุขภาพเชิงพาณิชย์ หนึ่งในประเด็นสำคัญคือ ความโปร่งใสและการอธิบายผล (interpretability) ของโมเดล AI ซึ่งหน่วยงานกำกับดูแล (เช่น FDA/EMA) และคณะกรรมการจริยธรรมมักให้ความสำคัญ เนื่องจากการตัดสินใจทางคลินิกที่อ้างอิงจาก "กล่องดำ" อาจทำให้ยากต่อการประเมินความปลอดภัยและประสิทธิผลของสารใหม่ งานศึกษาหลายชิ้นและแบบสำรวจในอุตสาหกรรมชี้ว่า ราว 60–80% ของกรรมการประเมินโครงการวิจัยให้ความสำคัญกับการอธิบายผลเป็นปัจจัยในการอนุญาตการทดลองทางคลินิก

ประเด็นด้านความเป็นส่วนตัวของข้อมูลผู้ป่วยเป็นอีกเรื่องที่มีน้ำหนัก ข้อมูลจีโนมิกและเรคคอร์ดทางการแพทย์ที่ใช้ฝึกโมเดลมีความละเอียดอ่อนและเสี่ยงต่อการระบุตัวตน การปฏิบัติที่แนะนำได้แก่การเข้ารหัสข้อมูลทั้งขณะส่งและขณะเก็บ การใช้เทคโนโลยีเช่น homomorphic encryption ที่อนุญาตให้ประมวลผลข้อมูลที่เข้ารหัสได้ และ federated learning ที่เก็บข้อมูลไว้ที่แหล่งต้นทางแล้วรวมผลการเรียนรู้โดยไม่ต้องย้ายข้อมูลดิบออกจากโรงพยาบาลหรือคลินิก นอกจากนี้การนำเทคนิคเช่น differential privacy มาปรับใช้สามารถลดความเสี่ยงของการเปิดเผยข้อมูลส่วนบุคคลจากโมเดลที่เผยแพร่หรือให้บริการทางคลินิก
ด้านทรัพย์สินทางปัญญา (IP) มีคำถามเชิงกฎหมายที่ยังไม่ชัดเจน เช่น ใครเป็นเจ้าของสิทธิบัตรของโมเลกุลที่ถูกออกแบบโดย AI—ผู้พัฒนาโมเดล บริษัทที่ลงทุน ข้อมูลต้นทาง หรือระบบ AI เอง กรณีที่มีการโต้แย้งเกี่ยวกับการเป็น "ผู้ประดิษฐ์" ของ AI (ตัวอย่างเช่นคดีที่เกี่ยวกับระบบ DABUS) ได้ยกประเด็นว่ากฎหมายสิทธิบัตรในหลายเขตอำนาจยังต้องการให้มีบุคคลธรรมดาเป็นผู้ประดิษฐ์ ซึ่งส่งผลต่อการยอมรับการยื่นขอสิทธิบัตรของโมเลกุลที่สร้างโดยระบบอัตโนมัติ ในทางปฏิบัติ ภาคธุรกิจควรจัดทำข้อตกลงด้านสิทธิระหว่างผู้มีส่วนร่วมทั้งหมด (เช่น สถาบันวิจัย ผู้ให้ข้อมูล เจ้าของโมเดล) และพิจารณารูปแบบการคุ้มครองที่หลากหลาย เช่น สิทธิบัตร การคุ้มครองความลับทางการค้า หรือสัญญาอนุญาตใช้งาน
หน่วยงานกำกับดูแลเช่น FDA และ EMA กำลังพัฒนาแนวทางและแผนการประเมินสำหรับผลิตภัณฑ์ที่อาศัย AI/ML (ตัวอย่างเช่น FDA AI/ML Action Plan) ซึ่งเน้นเรื่องการตรวจสอบวงจรชีวิตของโมเดล การควบคุมคุณภาพข้อมูล การทวนสอบผลและระบบการติดตามหลังการออกสู่ตลาด (post-market surveillance) สำหรับผู้ประกอบการ แนวทางปฏิบัติที่ดีควรรวมถึง:
- การบันทึกและเอกสารครบถ้วน — เก็บ audit trail ของชุดข้อมูล กระบวนการฝึกโมเดล และการปรับปรุงรุ่น (model versioning) เพื่อสนับสนุนการยื่นขอและการตรวจสอบ
- การอธิบายผลที่รองรับการตัดสินใจทางคลินิก — ใช้เทคนิค Explainable AI, model cards และการทดสอบเชิงสาเหตุ (causal validation) เพื่อสื่อสารความเสี่ยงและขอบเขตการใช้งาน
- การคุ้มครองข้อมูลและการปฏิบัติตามกฎระเบียบ — นำมาตรการเข้ารหัส การยินยอมจากผู้ป่วย การจัดการสิทธิ์เข้าถึง และการประเมินผลกระทบด้านความเป็นส่วนตัว (DPIA) มาใช้
- การจัดการ IP ที่ชัดเจน — ระบุความเป็นเจ้าของก่อนเริ่มโครงการ กำหนดนโยบายการแบ่งปันผลประโยชน์ และเตรียมกลยุทธ์การยื่นขอสิทธิบัตรหรือความคุ้มครองอื่น ๆ
- การมีปฏิสัมพันธ์เชิงรุกกับหน่วยงานกำกับ — ประสานงานกับผู้ตรวจสอบตั้งแต่ต้นโครงการ เพื่อให้การออกแบบการทดลองและเอกสารสนับสนุนสอดคล้องกับความคาดหวังของผู้กำกับดูแล
สรุปแล้ว การนำ Machine Learning มาร่วมในกระบวนการค้นพบยาเป็นความก้าวหน้าทางวิทยาศาสตร์ที่สำคัญ แต่เพื่อให้เกิดประโยชน์อย่างยั่งยืนและปลอดภัย องค์กรต้องให้ความสำคัญกับความโปร่งใสของโมเดล การปกป้องข้อมูลผู้ป่วย การจัดการสิทธิในทรัพย์สินทางปัญญา และการปฏิบัติตามแนวทางของหน่วยงานกำกับดูแลอย่างเป็นระบบและโปรแอคทีฟ
อนาคตและข้อเสนอแนะ: แนวโน้ม ผลกระทบต่อระบบสาธารณสุข และคำแนะนำต่อองค์กร
แนวโน้มใน 5–10 ปีข้างหน้า
ในช่วง 5–10 ปีข้างหน้า เราคาดว่าเทคโนโลยี Machine Learning (ML) จะผสานกับระบบอัตโนมัติในห้องปฏิบัติการจนเกิดเป็นแนวทาง closed-loop discovery ที่สามารถออกแบบ ทดสอบ และเรียนรู้จากผลการทดลองได้โดยอัตโนมัติ แบบวนรอบ (design–build–test–learn) ตัวอย่างปัจจุบันจากบริษัทเช่น Recursion และโครงการห้องปฏิบัติการหุ่นยนต์ชี้ให้เห็นศักยภาพในการทำงานแบบนี้ โดยคาดว่าแนวทางดังกล่าวอาจช่วยลดเวลาสำหรับการค้นพบสารนำ (lead identification) ได้ประมาณ 20–40% และเพิ่มประสิทธิภาพการคัดกรองสารตั้งต้นอย่างมีนัยสำคัญ
ควบคู่ไปกับ closed-loop discovery จะเป็นการเติบโตของ personalized medicine driven by ML ที่ใช้ข้อมูลจีโนม ข้อมูลคลินิก และข้อมูลพฤติกรรมเพื่อออกแบบการรักษาเฉพาะบุคคลมากขึ้น ML จะช่วยระบุ biomarker ที่เกี่ยวข้องกับการตอบสนองยา ทำให้สามารถแบ่งกลุ่มผู้ป่วยให้แคบลงและเพิ่มอัตราความสำเร็จของการทดลองทางคลินิกได้ คาดว่าอัตราความสำเร็จของโมเลกุลที่คัดเลือกด้วย ML และตรวจกำหนดเป้าหมายดีขึ้นอาจเพิ่มขึ้นได้ในระดับ สองหลัก (%) เมื่อเทียบกับวิธีดั้งเดิม
การใช้ real-world evidence (RWE) จะกลายเป็นส่วนสำคัญของระบบนิเวศการค้นพบยา โดยข้อมูลจากระบบดูแลสุขภาพจริง (EHR, claims, registry และอุปกรณ์สวมใส่) จะถูกนำมาเติมเต็มข้อมูลจากการทดลองเชิงควบคุมเพื่อประเมินประสิทธิผลและความปลอดภัยในประชากรที่หลากหลาย กรอบการนำ RWE มาใช้ได้รับการสนับสนุนจากหน่วยงานกำกับ เช่น FDA ที่ประกาศแนวทางการใช้ RWE ซึ่งช่วยเปิดทางให้การอนุมัติและการประเมินมูลค่าทางเศรษฐกิจของยาเปลี่ยนแปลงไป
ผลกระทบต่อระบบสาธารณสุข
ทิศทางดังกล่าวจะส่งผลเชิงบวกต่อความเร็วและความเฉพาะทางของการรักษา โดยทำให้การค้นพบและการปรับใช้ยามีความรวดเร็วขึ้น ลดเวลาและต้นทุนของการพัฒนา และเพิ่มโอกาสในการพัฒนายาสำหรับโรคหายาก (orphan diseases) ซึ่งเดิมอาจไม่ได้รับการลงทุนเพียงพอ ตัวอย่างคือโมเดลธุรกิจแบบ platform-as-a-service หรือ licensing ของแพลตฟอร์ม ML จะช่วยให้บริษัทขนาดเล็กสามารถเข้าถึงความสามารถด้านการออกแบบยาได้มากขึ้น
อย่างไรก็ตาม ต้องให้ความสำคัญกับ ความเท่าเทียมในการเข้าถึง เทคโนโลยีและยาที่เกิดจาก ML ความเสี่ยงที่เกิดขึ้นได้แก่ การกระจุกตัวของทรัพยากร (data, computing, ความเชี่ยวชาญ) ในประเทศหรือองค์กรที่มีศักยภาพสูง ซึ่งอาจนำไปสู่ความเหลื่อมล้ำในการเข้าถึงการรักษา นอกจากนี้ยังมีความเสี่ยงด้านความเป็นส่วนตัวและความน่าเชื่อถือของข้อมูล RWE ซึ่งหากไม่ได้มาตรฐานอาจส่งผลต่อความปลอดภัยของผู้ป่วย
คำแนะนำเชิงปฏิบัติสำหรับนักวิจัย ภาคอุตสาหกรรม และผู้กำกับดูแล
- ลงทุนในโครงสร้างพื้นฐานข้อมูล — องค์กรควรลงทุนในระบบจัดเก็บข้อมูลที่ปลอดภัย พร้อมมาตรฐานเชิงสัญลักษณ์ (interoperability) เช่น OMOP, FHIR และการใช้ ontology ร่วมกัน เพื่อให้ RWE และข้อมูลทดลองสามารถใช้งานร่วมกันได้อย่างมีประสิทธิภาพ
- สร้างทีมผสม (interdisciplinary) — การพัฒนายาที่ขับเคลื่อนด้วย ML ต้องการทีมที่ผสมผสานนักวิทยาศาสตร์ข้อมูล วิศวกรซอฟต์แวร์ นักชีววิทยา แพทย์ และผู้เชี่ยวชาญด้านกฎระเบียบ การลงทุนในการฝึกอบรมข้ามสาขาและการดึงผู้เชี่ยวชาญจากหลายโดเมนจะเป็นกุญแจสำคัญ
- ทำงานใกล้ชิดกับหน่วยงานกำกับ — ควรร่วมมือกับหน่วยงานกำกับเพื่อพัฒนามาตรฐานการประเมินโมเดล ML, โปรโตคอลการยืนยันผล และกรอบการใช้ RWE การทำ pilot studies ที่มีการเปิดเผยวิธีการและผลลัพธ์สามารถช่วยสร้างความเชื่อมั่นและลดอุปสรรคด้านการอนุญาต
- ส่งเสริมความเป็นธรรมและการเข้าถึง — องค์กรควรออกแบบแผนการเข้าถึงทางการเงิน เช่น ราคาตามมูลค่า (value-based pricing), การแบ่งปันเทคโนโลยีในรูปแบบ precompetitive commons และโครงการความร่วมมือระหว่างประเทศเพื่อป้องกันการกระจุกตัวของประโยชน์
- วางกรอบจริยธรรมและความปลอดภัยของข้อมูล — พัฒนานโยบายความเป็นส่วนตัว การยินยอมตามมาตรฐาน และการทดสอบความเอนเอียงของโมเดล (bias audits) เป็นกระบวนการต่อเนื่องเพื่อให้การตัดสินใจของ ML โปร่งใสและยุติธรรม
- เริ่มจากโครงการนำร่องและการวัดผล — แนะนำให้เริ่มด้วยโครงการนำร่องที่มีขอบเขตชัดเจน วัดผลด้วยตัวชี้วัดเชิงคุณภาพและเชิงปริมาณ (เช่น เวลาในการค้นพบ, อัตราความสำเร็จของการทดลอง, ผลลัพธ์ด้านผู้ป่วย) เพื่อนำบทเรียนมาปรับใช้แบบขยายผล
โดยสรุป การผสาน ML กับกระบวนการค้นพบยาเป็นโอกาสสำคัญที่จะยกระดับนวัตกรรมทางการแพทย์ แต่ความสำเร็จเชิงระบบขึ้นอยู่กับการลงทุนในโครงสร้างพื้นฐานข้อมูล การสร้างกำลังคนแบบข้ามสาขา และความร่วมมือกับหน่วยงานกำกับเพื่อกำหนดมาตรฐานร่วมที่คำนึงถึงความเท่าเทียมและความปลอดภัยของผู้ป่วย การลงมือเชิงปฏิบัติในตอนนี้จะช่วยให้ภาคสุขภาพสามารถเก็บเกี่ยวผลประโยชน์จากการปฏิวัติครั้งนี้ได้อย่างยั่งยืน
บทสรุป
การประยุกต์ใช้ Machine Learning (ML) ในการค้นพบยาได้เปลี่ยนรูปแบบวงการแพทย์อย่างรวดเร็ว โดยช่วยเร่งกระบวนการค้นหาและออกแบบโมเลกุล ลดเวลาและต้นทุนในการค้นพบเบื้องต้น และขยายขอบเขตของแนวทางการออกแบบยา ตัวอย่างเช่น กรณีศึกษาจากบริษัทบางแห่งรายงานความสามารถในการระบุโมเลกุลนำร่องภายในระยะเวลาหลายสัปดาห์ถึงหลายเดือน และงานวิจัยเชิงเปรียบเทียบชี้ว่าการใช้เทคนิคเชิงคำนวณร่วมกับข้อมูลขนาดใหญ่สามารถลดเวลาในขั้นตอนค้นพบได้อย่างมีนัยสำคัญ (ในบางกรณีลดลงเป็นหลักสิบเปอร์เซ็นต์ถึงมากกว่า 50%) อย่างไรก็ดี ML ไม่ได้มาแทนที่การทดลองเชิงชีวภาพ—ผลลัพธ์ที่เชื่อถือได้ที่สุดมาจากการผสานระหว่างโมเดลเชิงคอมพิวเตอร์และการทดลองในห้องปฏิบัติการจริง เช่น การใช้การพยากรณ์โครงสร้างโปรตีนเพื่อชี้เป้าการทดลองในห้องปฏิบัติการและยืนยันผลด้วยข้อมูลทางชีวภาพ
เพื่อให้ประโยชน์ของ ML ถูกนำไปใช้อย่างปลอดภัยและเป็นธรรม จำเป็นต้องให้ความสำคัญกับคุณภาพของข้อมูล ความโปร่งใสและการตรวจสอบได้ของโมเดล การปฏิบัติตามกฎระเบียบ รวมถึงการลงทุนในโครงสร้างพื้นฐานข้อมูลและพัฒนาทักษะของบุคลากร (data engineering, ML ops, ภาระงานทางคลินิกและกฎระเบียบ) เท่านั้นการรวมกันของนโยบาย ดัชนีคุณภาพข้อมูล มาตรฐานการอธิบายผล และการร่วมมือระหว่างนักวิจัย ภาคอุตสาหกรรม และหน่วยงานกำกับดูแล จะทำให้เทคโนโลยีนี้นำไปสู่การรักษาที่มีประสิทธิภาพ ปลอดภัย และเข้าถึงได้มากขึ้นในอนาคต โดยทิศทางต่อไปคือระบบปฏิบัติการแบบผสาน (hybrid workflows) ที่ผสาน ML กับการทดลองเชิงปฏิบัติและการประเมินทางคลินิกอย่างต่อเนื่องเพื่อเร่งการแปลผลงานวิจัยสู่การรักษาจริง
📰 แหล่งอ้างอิง: Technology Networks




